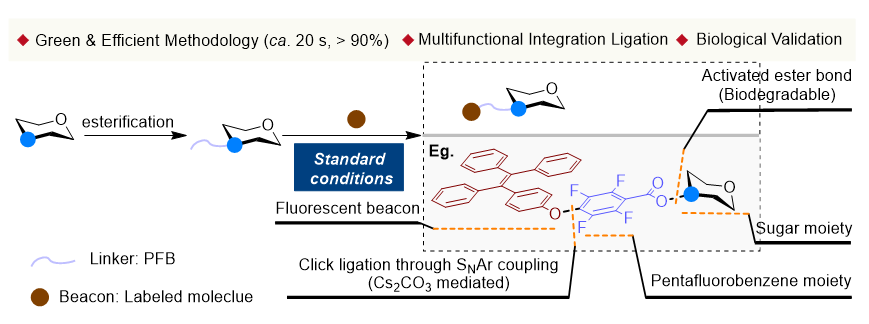
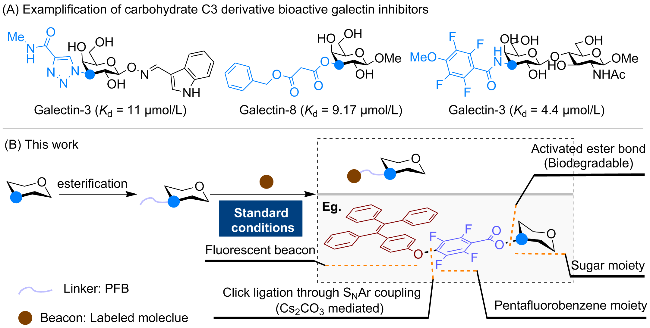
1 引言
2 结果与讨论
2.1 反应条件优化
表1 条件优化aTable 1 Reaction condition optimizationa |
| Entrya | Base | Solvent (V/V) | Time | Aa-1 Yieldc/% |
|---|---|---|---|---|
| 1 | Cs2CO3 | THF | 12 h | 33.8 |
| 2 | Cs2CO3 | DMSO | 12 h | 71.5 |
| 3 | Cs2CO3 | DMF | 12 h | 42.6 |
| 4 | Cs2CO3 | THF/DMF (5∶1) | 12 h | 67.3 |
| 5 | Cs2CO3 | THF/NMP (5∶1) | 12 h | 55.2 |
| 6 | Cs2CO3 | THF/DMSO (5∶1) | 12 h | 95.7 |
| 7 | DBU | THF/DMSO (5∶1) | 12 h | NR |
| 8 | K2CO3 | THF/DMSO (5∶1) | 12 h | 3.0 |
| 9 | NaHCO3 | THF/DMSO (5∶1) | 12 h | 14.0 |
| 10 | NaOAc | THF/DMSO (5∶1) | 12 h | 3.0 |
| 11 | Quinine | THF/DMSO (5∶1) | 12 h | 60.7 |
| 12b | — | THF/DMSO (5∶1) | 12 h | NR |
| 13d | Cs2CO3 | THF/DMSO (5∶1) | 20 s | 92.9 |
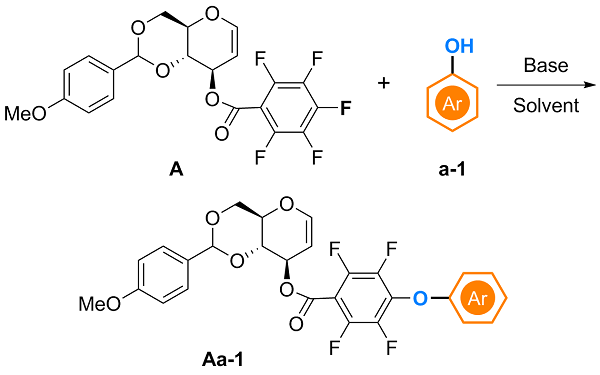
a Pentafluorobenzoylated PMPGlucal A (1.0 equiv.), phenol a-1 (1.1 equiv.), and Cs2CO3 (1.0 equiv.) were added to a 15 mL reaction tube. The reaction solvent (0.1 mmol/L) was charged, and the mixture was reacted at room temperature under rapid stirring for 20 s to 12 h. For the purpose of emphasis, in this article, Ar was used to represent a benzene ring, and similarly hereinafter. b No base was employed in the reaction, NR: no reaction. c The target compound was detected with isolated yields. d The reaction time was shorten to 20 s under standard conditions. THF: tetrahydrofuran; DMF: N,N-dimethyl- formamide; NMP: N-methylpyrrolidone. |
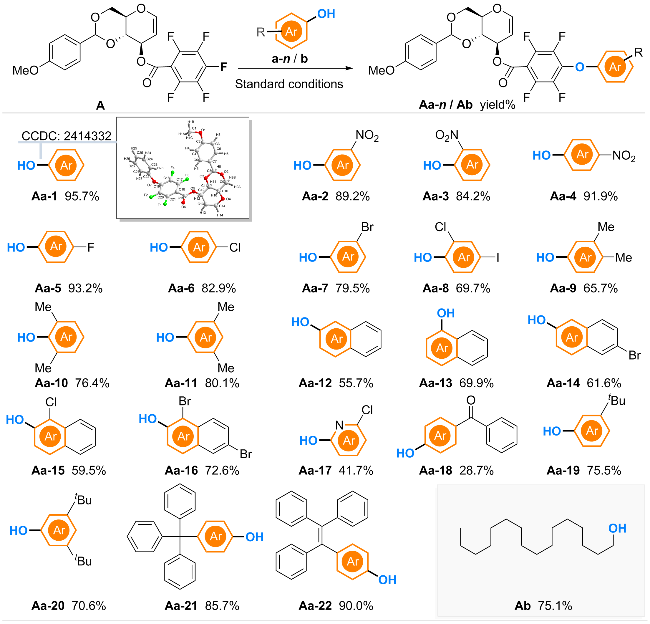
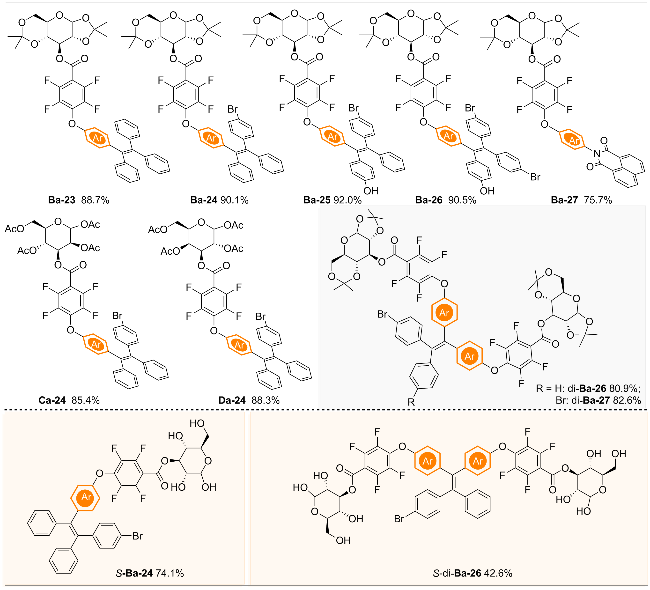
2.2 底物拓展
2.3 通用糖单元C3功能衍生构建及识别应用探索
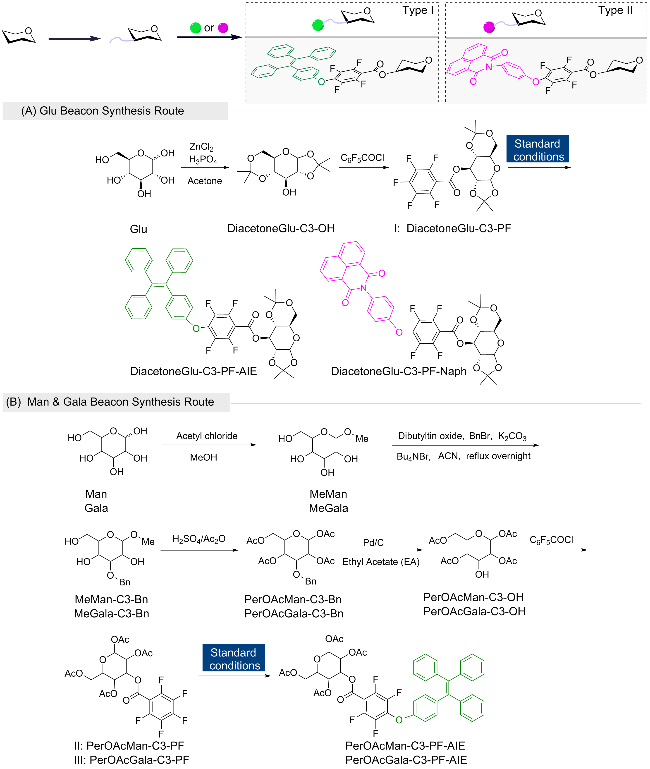
图3 通用糖分子C3位点衍生化及合成路线: (A) Glu C3 beacon合成路线设计; (B) Man & Gala C3 beacon合成路线设计Figure 3 Derivatization and synthetic routes of general sugar molecules at the C3 position: (A) Synthetic route design of Glu C3 beacon; (B) Synthetic route design of Man & Gala C3 beacons |
图5 葡萄糖C3聚集诱导发光分子在PBS缓冲液(10 mmol/L, 1 mg/mL CaCl2、1 mg/mL Mn(NO3)2, pH 7.04)中与蛋白的响应特性. (A) λex=257 nm, λem=467 nm条件下, S-Ba-24 (2.5 μmol/L, PBS)与不同浓度Con A蛋白(0~0.2 μmol/L)作用下荧光强度变化趋势(2.2倍增强); (B)相同条件下, S-Ba-24 (2.5 μmol/L, PBS)与不同浓度PNA蛋白(0~0.2 μmol/L)作用下荧光强度变化趋势(1.4倍增强); (C) S-di-Ba-26 (2.5 μmol/L, PBS)与不同浓度Con A蛋白(0~0.2 μmol/L)作用下荧光强度变化趋势(3.1倍增强)Figure 5 Response of glucose C3 aggregation-induced emission (AIE) molecules with proteins in PBS buffer (10 mmol/L, containing 1 mg/mL CaCl2, 1 mg/mL Mn(NO3)2, pH 7.04). (A) Fluorescence intensity changes (2.2-fold enhancement) of S-Ba-24 (2.5 μmol/L, PBS) upon interaction with different concentrations (0~0.2 μmol/L) of Con A (λex=257 nm, λem=467 nm); (B) Fluorescence intensity changes (1.4-fold enhancement) of S-Ba-24 (2.5 μmol/L, PBS) upon interaction with different concentrations (0~0.2 μmol/L) of PNA; (C) Fluorescence intensity changes (3.1-fold enhancement) of S-di-Ba-26 (2.5 μmol/L, PBS) upon interaction with different concentrations (0~0.2 μmol/L) of Con A |