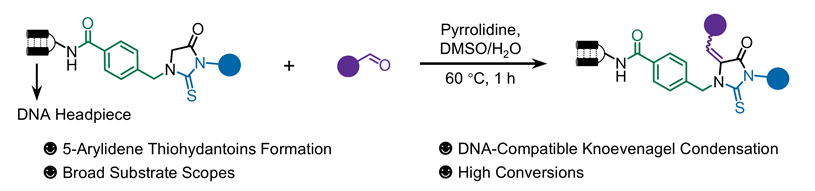
1 引言
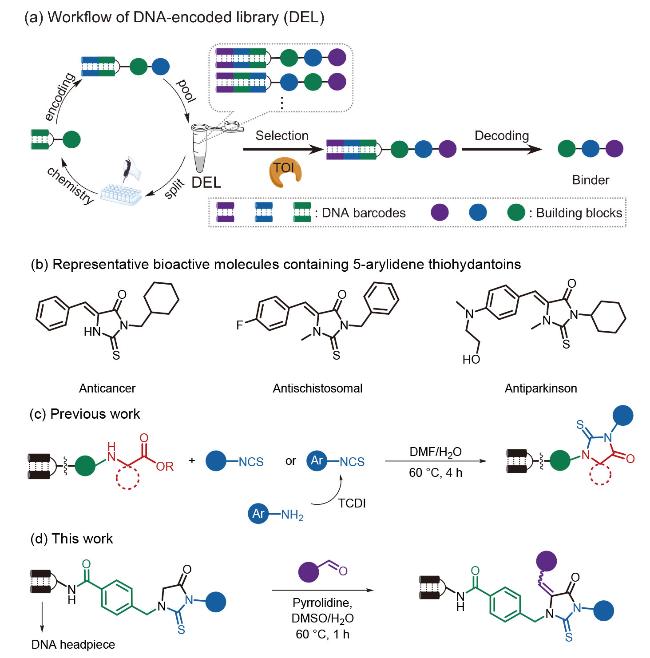
图1 (a) DNA编码分子库的工作流程. (b)含有5-亚芳基硫代乙内酰脲的代表性生物活性分子. (c)前期的工作. (d)通过Knoevenagel缩合反应on-DNA合成5-亚芳基硫代乙内酰脲Figure 1 (a) Workflow of DNA-encoded library (DEL). (b) Representative bioactive molecules containing 5-arylidene thiohydantoins. (c) Previous work. (d) On-DNA synthesis of 5-arylidene thiohydantoins via Knoevenagel condensation |
2 结果与讨论
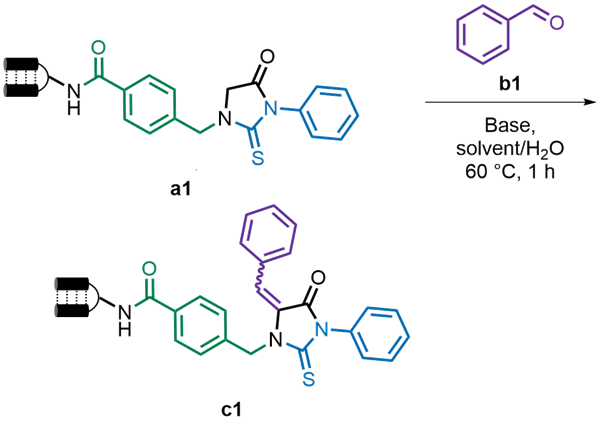
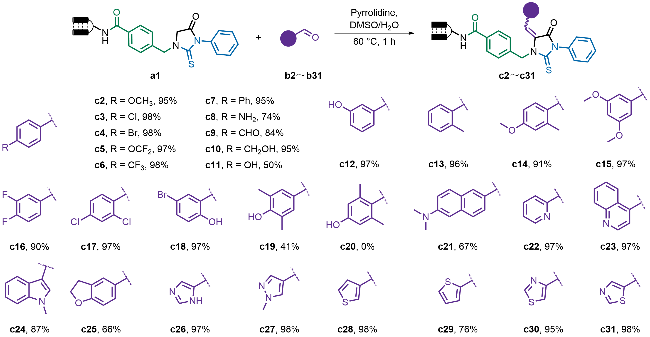
2.1 条件筛选
表1 反应条件筛选aTable 1 Optimization of reaction conditionsa |
| Entry | Base | Solvent | Conversionb/% |
|---|---|---|---|
| 1 | Morpholine | DMA | 18 |
| 2 | Et3N | DMA | 5 |
| 3 | DBU | DMA | 0 |
| 4 | PMP | DMA | 0 |
| 5 | PMDETA | DMA | 10 |
| 6 | DABCO | DMA | 5 |
| 7 | HTMP | DMA | 8 |
| 8 | Piperidine | DMA | 50 |
| 9 | Me6TREN | DMA | 12 |
| 10 | Pyrrolidine | DMA | 80 |
| 11 | — | DMA | 0 |
| 12 | Pyrrolidine | EtOH | 80 |
| 13 | Pyrrolidine | MeOH | 85 |
| 14 | Pyrrolidine | ACN | 76 |
| 15 | Pyrrolidine | DMF | 90 |
| 16 | Pyrrolidine | DMSO | 97 |
a Conditions: DNA conjugate a1 (2 μL, 100 μmol/L in H2O, 0.2 nmol), solvent (3 μL), H2O (8 μL), benzaldehyde b1 (5 μL, 500 mmol/L in solvent), base (2 μL, 200 mmol/L in H2O), 60 ℃, 1 h; b Conversions were determined by UHPLC-MS (Ultra-high-performance liquid chromatography tandem mass spectrometry). DMA: N,N-dimethylacetamide; DBU: 1,8-diazabicyclo[5.4.0]- undecane-7-ene; PMP: 1,2,2,6,6-pentamethyl-4-piperidinol; PMDETA: pentamethyldiethylenetriamine; DABCO: 1,4-diazobicyclo[2.2.2]octane; HTMP: 2,2,6,6-tetramethylpiperidine; Me6TREN: tris(2-dimethylaminoethyl)amine; ACN: acetonitrile; DMF: N,N-dimethylformamide. |