1 引言
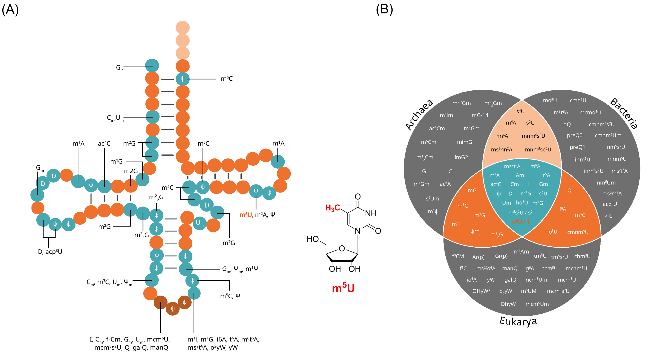
2 m5U的检测及其修饰酶
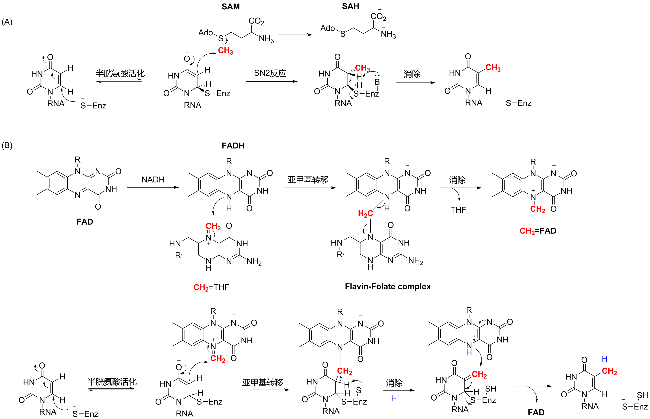
图2 不同物种中m5U的形成机制. (A)以SAM为甲基供体的酶催化机制(哺乳动物细胞、大肠杆菌等); (B)以N5,N10-亚甲基四氢叶酸为甲基供体的酶催化机制(部分革兰氏阳性菌如: 枯草芽孢杆菌及支原体等)Figure 2 Mechanism of m5U formation in different species. (A) Enzyme catalytic mechanism using SAM as a methyl donor (mammalian cells, E. coli); (B) Enzyme catalytic mechanism using N5,N10-methylenetetrahydrofolate as a methyl donor (Some gram positive bacteria, such as B. subtilis and mycoplasma) |
3 m5U的生物学功能
3.1 m5U提升tRNA的稳定性
3.2 m5U与其他修饰的互作
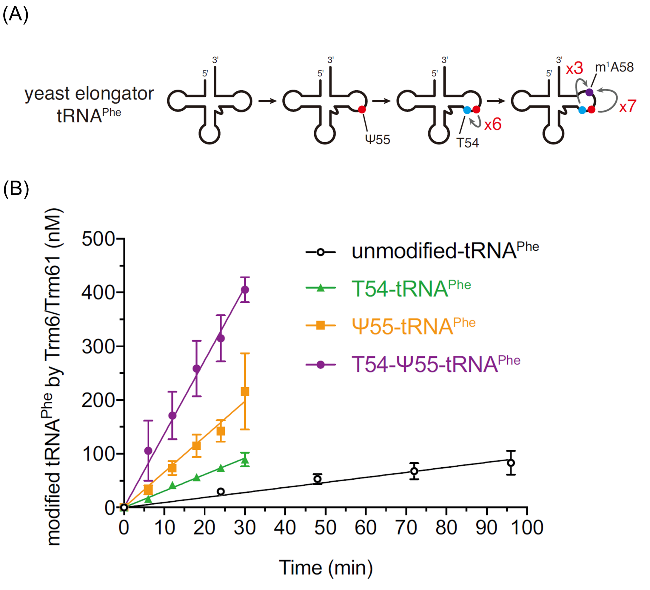
图3 Ψ55、m5U54及m1A58之间的互作. (A)Ψ55、m5U54及m1A58引入的先后顺序; (B)Trm6/Trm61催化含有Ψ55、m5U54的tRNA A58甲基化的速率差别. 图3来自参考文献[21]Figure 3 Figure 3 Cross-talk between Ψ55, m5U54 and m1A58. (A) The introduce order of Ψ55, m5U54 and m1A58 in tRNA. (B) The methylation rate of tRNA A58 containing Ψ55 or m5U54 catalyzed by Trm6/Trm61. was adapted from ref. [21]. Available under a CC-BY license. Copyright 2023, The Authors |
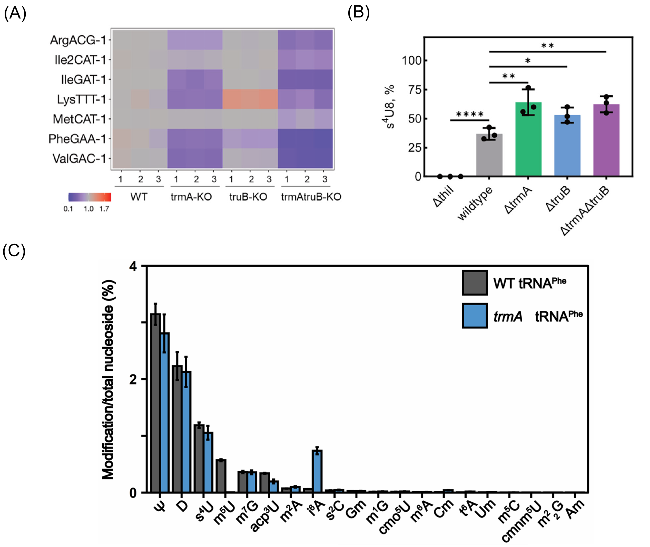
图4 tRNA m5U54与其它修饰的互作. (A)敲除trmA、truB后大肠杆菌tRNA acp3U47变化的热图; (B) S4U8的变化; (C)敲除trmA后大肠杆菌tRNA上的修饰变化(通过LC-MS/MS定量). 图4A、 4B来自参考文献[24], 图4C来自参考文献[25]Figure 4 Cross-talk between m5U54 and other modifications in tRNA. (A) Heatmap depicting the variations in Escherichia coli tRNA acp3U47 following the knockout of trmA and truB; (B) changes of S4U8; (C) Modification changes of E. coli tRNA afterΔtrmA. Figures 4A, 4B were adapted from ref. [24]. Available under a CC-BY-NC-ND 4.0 license. Copyright 2024, The Authors; Figure 4C was adapted from ref. [25]. Available under a CC-BY-NC-ND 4.0 license. Copyright 2024, The Authors |
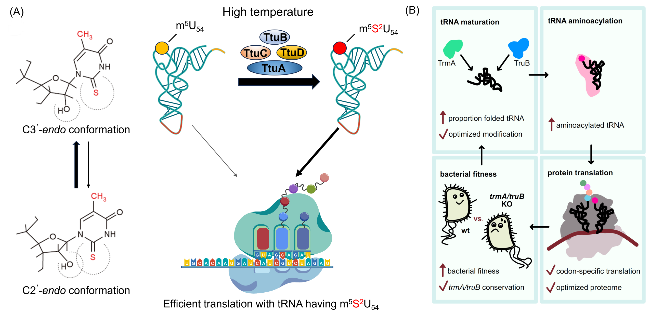
3.3 m5U调控翻译过程与细胞适应性
4 m5U甲基转移酶的多功能性
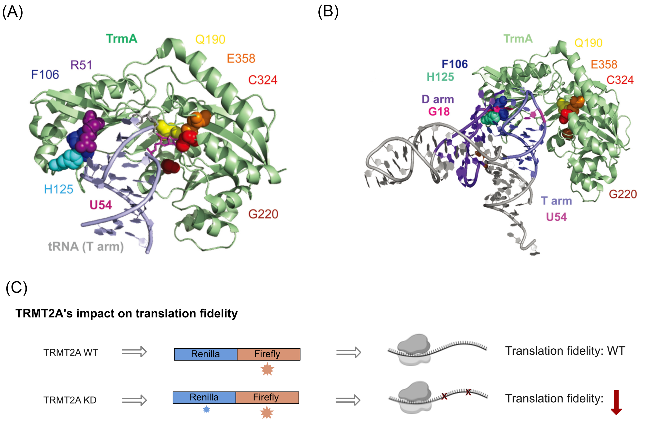
图6 (A) TrmA和tRNA的T臂结合的结构(PDB ID: 3BT7); (B) TrmA RNA结合结构域中F106-H125残基与tRNA D臂和T臂相互作用的结构示意图; (C) TRMT2A对翻译保真度的影响. 图6A、6B来自参考文献[31], 图6C来自参考文献[32]Figure 6 (A) Structure of TrmA bound to T arm of tRNA (PDB ID: 3BT7); (B) Structural model for the disruption of D arm and T arm tertiary interactions in tRNA by the F106-H125 wedge in the RNA-binding domain of TrmA; (C) TRMT2A’s impact on translation fidelity. Figure 6A, 6B were adapted from ref. [31]. Available under a CC-BY-NC license. Copyright 2020, The Authors; Figure 6C was adapted from ref. [32]. Available under a CC-BY license. Copyright 2023, The Authors |