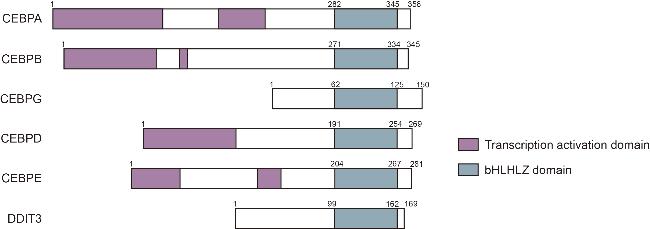
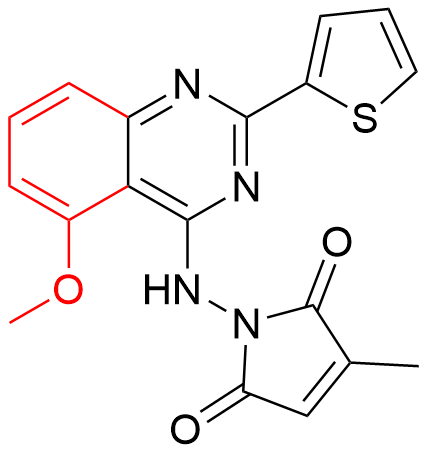
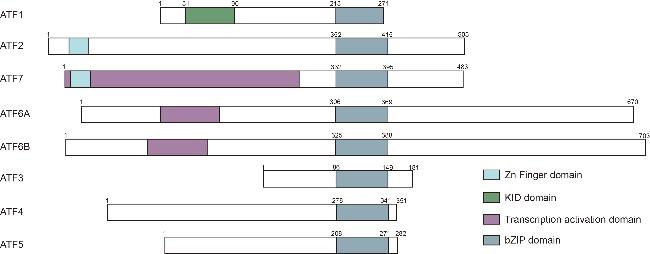
1 亮氨酸拉链转录因子的功能域
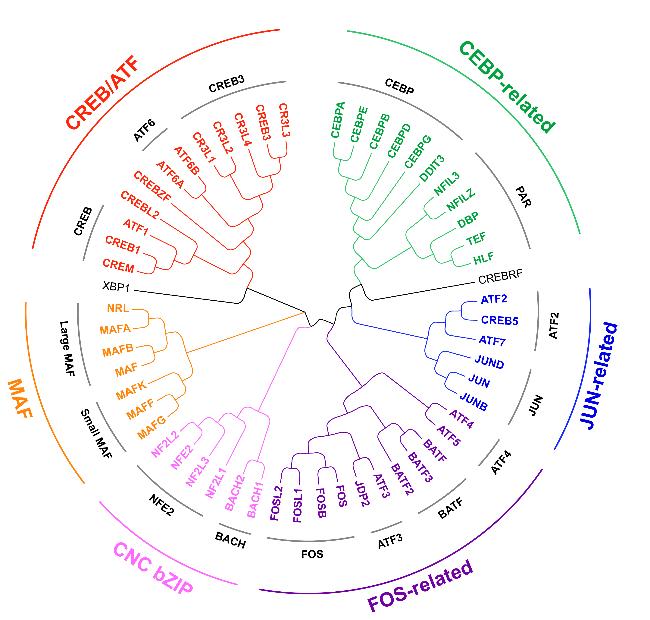
图1 人类基因组bZIP转录因子家族进化树, 由MEGA软件生成. 红色字为CREB/ATF家族. 绿色字为CEBP相关家族. 蓝色字为JUN相关家族. 紫色字为FOS相关家族. 淡紫色为CNC bZIP家族. 橙色字为MAF家族. 圆弧上黑色字为亚家族分类. 转录因子黑色字为独立分类.Figure 1 Likelihood tree of bZIP transcription factors in the human genome, built by MEGA. CREB/ATF transcription factors in red. CEBP-related transcription factors in green. JUN-related transcription factors in blue. FOS-related transcription factors in purple. CNC bZIP transcription factors in light purple. MAF transcription factors in orange. Subfamilies are classified in black on the arc. XRP1 and CREBRF are classified independently. |
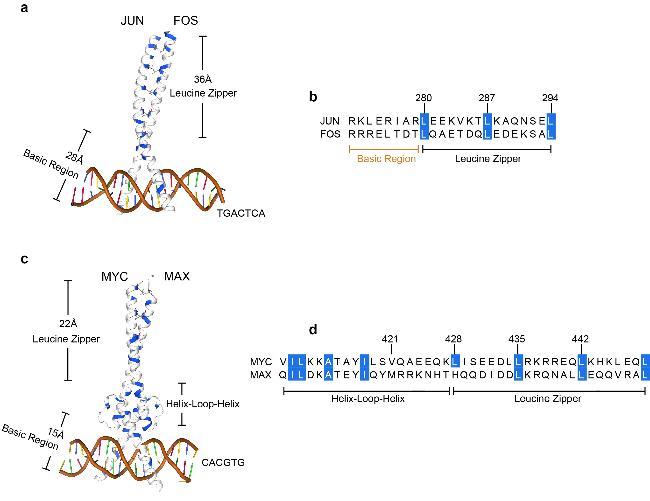
图2 bZIP转录因子JUN和FOS的二聚体复合物; bHLH-LZ转录因子MYC和MAX的二聚体复合物. (a) JUN和FOS的二聚体结合DNA的晶体结构(PDB: 1fos)[6]. JUN的bZIP区域(252aa~315aa)由64个氨基酸构成, 其中碱性区域占28个, LZ区域占36个. FOS的bZIP区域(137aa~220aa)长度均与JUN一致. 疏水氨基酸用蓝色标注. 碱性区域直接与DNA(红色)相互作用, 识别5'-TGACTCA-3'基序. (b) JUN和FOS的碱性区域与LZ区域连接处的氨基酸构成. 蓝色标注为符合拉链性质的疏水氨基酸位点. (c) MYC和MAX的二聚体结合DNA的晶体结构(PDB: 1nkp)[7]. MYC的bHLH-LZ区域(369aa~449aa)由81个氨基酸构成, 其中碱性区域占15个, HLH区域占44个, LZ区域占22个. MAX的bHLH-LZ区域(23aa~102aa)由80个氨基酸构成, 其中碱性区域相比MYC缺少位于首位的一个疏水氨基酸, 其余长度与MYC一致. 疏水氨基酸用蓝色标注. 碱性区域直接与DNA(红色)相互作用, 识别5'-CACGTG-3'基序[7]. (d) MYC和MAX的HLH区域与LZ区域连接处的氨基酸构成. LZ区域蓝色标注为符合拉链性质的疏水氨基酸位点, HLH区域蓝色标注为疏水氨基酸. (a), (c)由pymol软件绘制. (b), (d)由MEGA软件绘制, 序列来自Uniprot.Figure 2 Dimeric complex of the bZIP transcription factor JUN and FOS; dimeric complex of the bHLH-LZ transcription factor MYC and MAX. (a) Crystal structure of the dimer-bound DNA of JUN and FOS (PDB: 1fos)[6]. The bZIP domain (252aa~315aa) of JUN consists of 64 amino acids, of which the basic region accounts for 28 and the LZ region for 36. The length of the bZIP domain (137aa~220aa) of FOS are all consistent with JUN. Hydrophobic amino acids are labeled in blue. The basic region interacts directly with DNA (red) and recognizes the 5'-TGACTCA-3' motif. (b) Amino acid composition of the basic region of JUN and FOS at the junction with the LZ region. Hydrophobic amino acid sites consistent with zipper properties are labeled in blue. (c) Crystal structure of the dimer-bound DNA of MYC and MAX (PDB: 1nkp)[7]. The bHLH-LZ domain (369aa~449aa) of MYC consists of 81 amino acids, of which 15 are basic, 44 are HLH, and 22 are LZ regions. The bHLH-LZ region of MAX (23aa~102aa) consists of 80 amino acids, of which the basic region lacks one hydrophobic amino acid located in the first position compared to MYC, and the rest of the length is consistent with MYC. Hydrophobic amino acids are labeled in blue. The basic region interacts directly with DNA (red) and recognizes the 5'-CACGTG-3' motif. (d) Amino acid composition of the HLH region of MYC and MAX at the junction with the LZ region. The LZ region is labelled in blue as a hydrophobic amino acid site consistent with zipper properties, and the HLH region is labelled in blue as a hydrophobic amino acid pair. (a), (c) Built by pymol. (b), (d) Built by MEGA with sequences from Uniprot. |
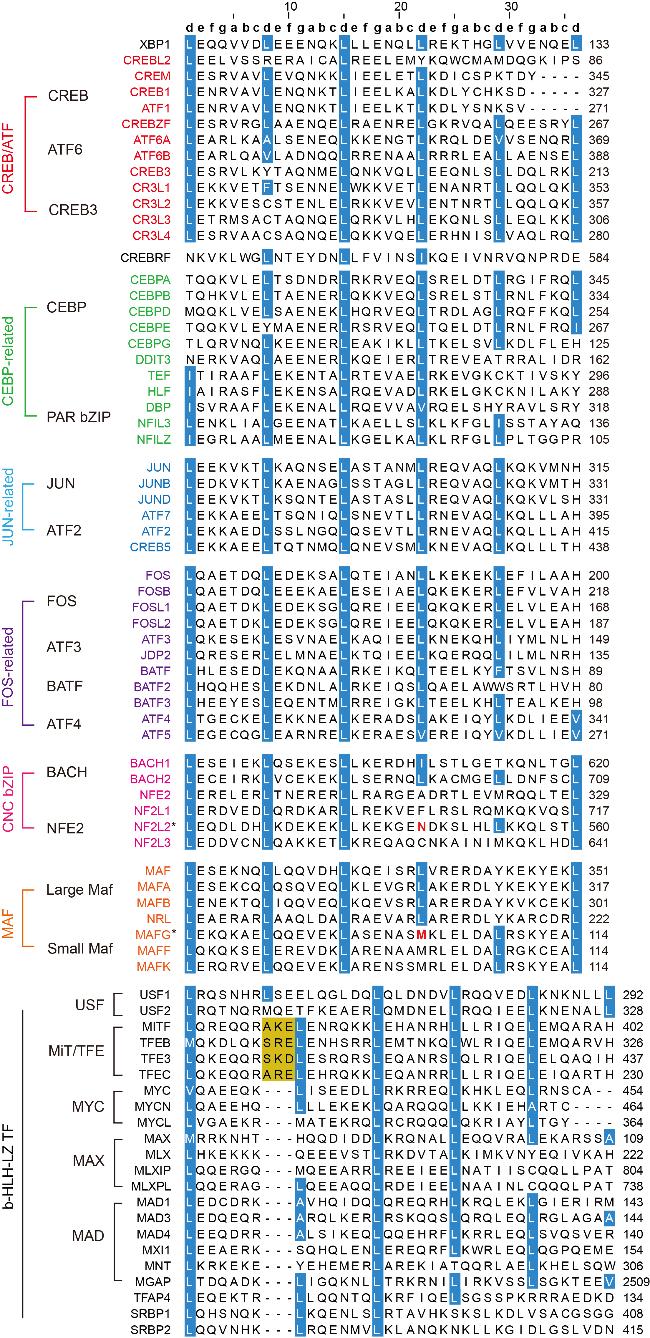
图3 人源LZ TF的亮氨酸拉链区域的氨基酸序列, 由MEGA软件绘制, 序列来自Uniprot. 蓝色标为各自转录因子家族中, 符合LZ循环规律且最具亲和度的疏水氨基酸. 黄色标为已经确定的氨基酸插入位置. *标和红色加粗字分别表示为NF2L2和MAFG的LZ序列和特定非疏水氨基酸位置.Figure 3 Amino acid sequence of the leucine zipper region of the human LZ TF, built by MEGA with sequences from Uniprot. The hydrophobic amino acids that conform to the LZ cycle pattern and have the highest affinity in the respective transcription factor family are labeled in blue. Identified amino acid insertion positions are labeled in yellow. *Highlighted and bolded red characters indicate the LZ sequence and specific non-hydrophobic amino acid positions for NF2L2 and MAFG, respectively. |
2 碱性亮氨酸拉链(basic Leucine zipper, bZIP)转录因子的功能及小分子调控
2.1 cAMP反应元件结合蛋白和激活转录因子蛋白(cAMP-Responsive Element-Binding Protein and Activating Transcription Factor, CREB/ATF)家族
2.1.1 cAMP反应元件结合蛋白(cAMP-Responsive Element-Binding Protein, CREB)亚家族
2.1.2 cAMP反应元件结合蛋白3 (cAMP-Responsive Element-Binding Protein 3, CREB3)亚家族
2.2 CCAAT/增强子结合蛋白相关家族(CCAAT/ Enhancer-Binding Protein-related, CEBP-related)
2.3 帽和领碱性亮氨酸拉链家族(Cap'n'Collar basic-region Leucine zipper, CNC bZIP)
2.3.1 BTB和CNC同源蛋白(BTB and CNC homolog, BACH)亚家族
2.3.2 转录因子NF-E2 45kDa亚基(transcription factor NF-E2 45 kDa subunit, NFE2)亚家族
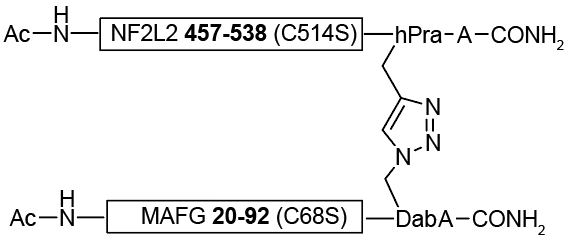
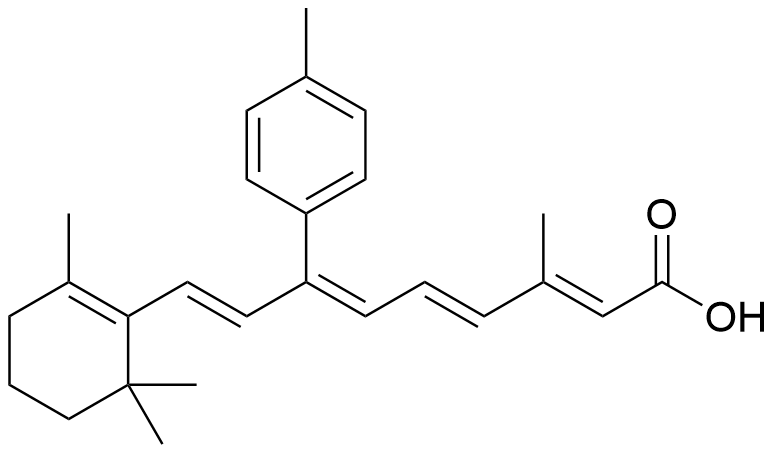
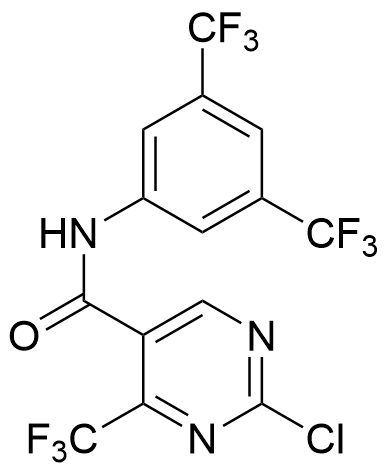
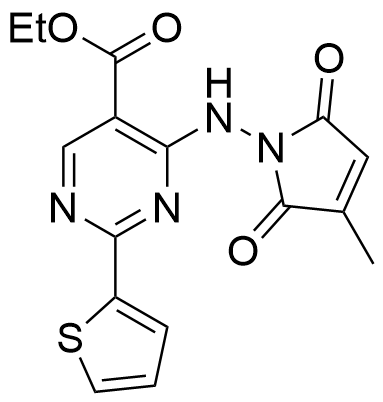
表3 直接靶向NF2L2的小分子/多肽抑制剂. 红色代表关键功能基团Table 3 Small molecule inhibitors or peptides directly targeting NF2L2. Red represent a critical functional group |
2.4 激活蛋白1 (Activator Protein 1, AP-1)转录因子
2.4.1 JUN (transcription factor JUN)亚家族
表4 直接靶向AP-1转录因子的小分子抑制剂. 蓝色代表CP1(3~7)位氨基酸. 红色代表关键功能基团Table 4 Small molecule inhibitors directly targeting AP-1 transcription factors. Blue represent amino acids at position CP1(3~7). Red represent a critical functional group |
2.4.2 激活转录因子(Activating transcription factor, ATF)(1~7)家族
3 碱性螺旋环螺旋-亮氨酸拉链(basic Helix-Loop-Helix Leucine zipper, bHLH-LZ)转录因子功能及小分子调控
3.1 小眼畸形/转录因子E家族(Microphthalmia/transcription factor E, MiT/TFE)
3.2 MYC原癌基因蛋白质家族(MYC proto-oncogene protein, MYC)
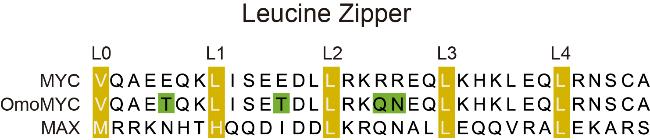
图8 OmoMYC与MYC和MAX序列对比. 图中黄色标为LZ区域的疏水氨基酸, 绿色标为OmoMYC与MYC相比, 氨基酸的具体突变位置[413].Figure 8 Comparison of OmoMYC with MYC and MAX sequences. The hydrophobic amino acids of LZ region are shown in yellow, and the specific amino acid mutation positions of OmoMYC compared to MYC are shown in green. |
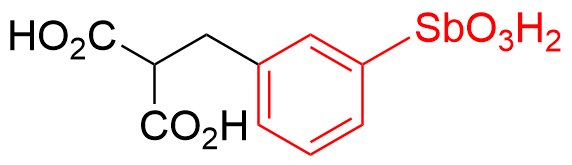
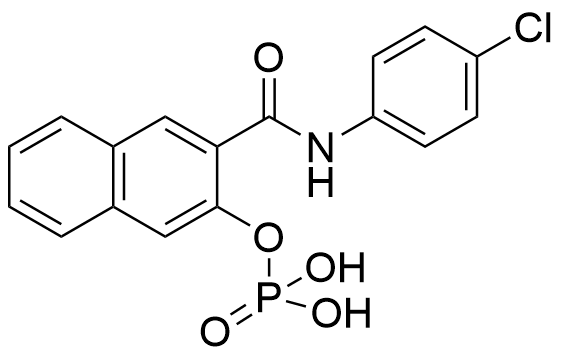
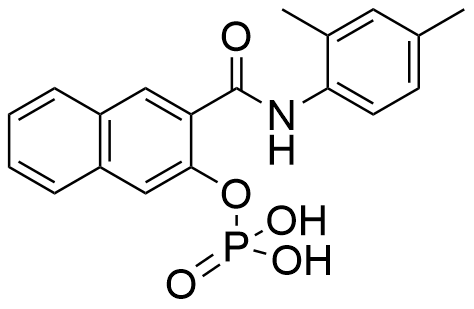
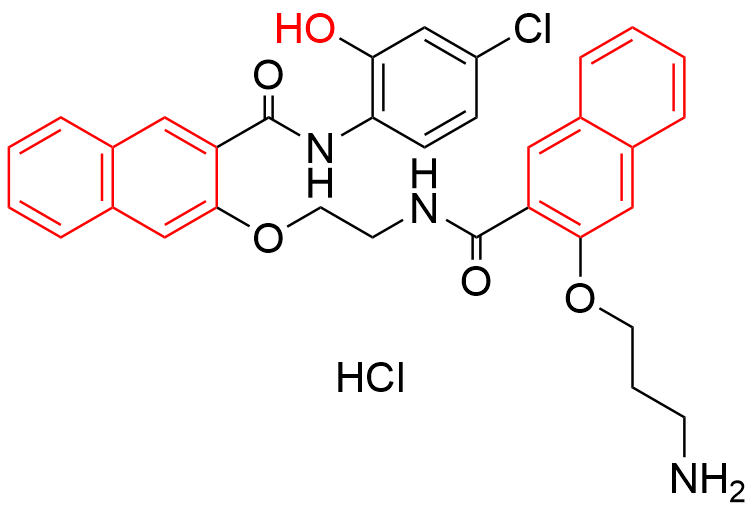
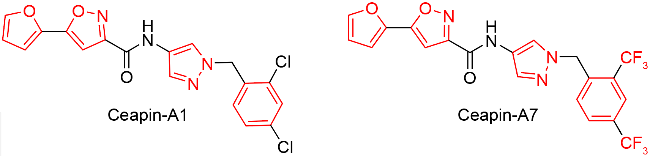
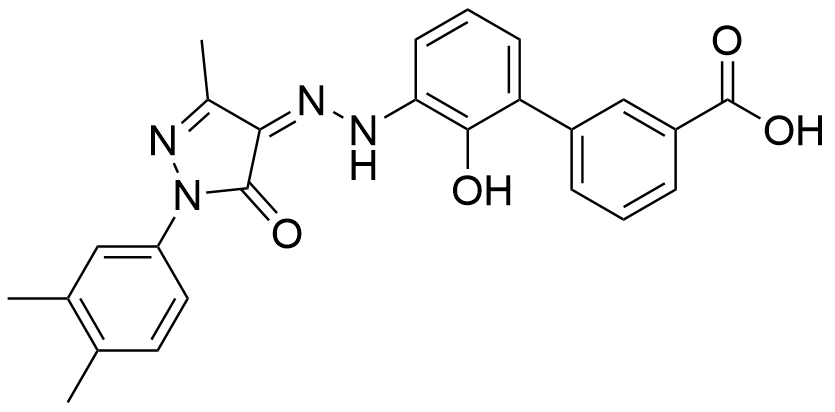
表6 直接靶向MYC的小分子抑制剂. 红色代表关键功能基团Table 6 Small molecule inhibitors directly targeting MYC. Red represent a critical functional group |
| Compound | Direct target | Mechanism |
|---|---|---|
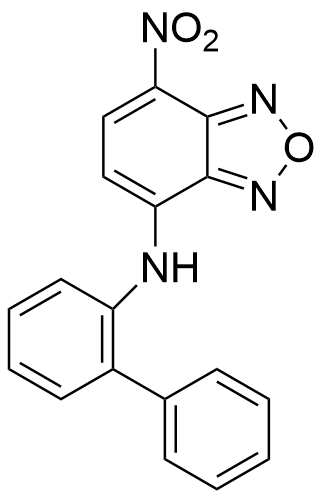 10074-G5[415-416] | MYC; MYC-MAX | 抑制MYC二聚体形成 |
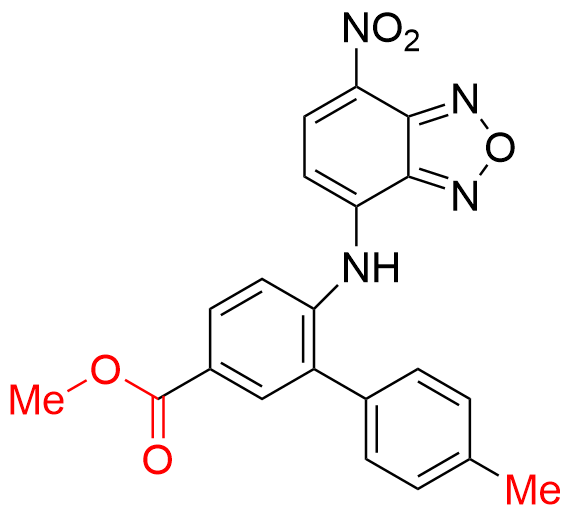 3jc48-3[425] | MYC-MAX | 抑制MYC二聚体形成 |
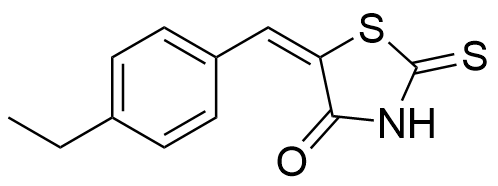 10058-F4 [415-416] | MYC; MYC-MAX | 抑制MYC二聚体形成 |
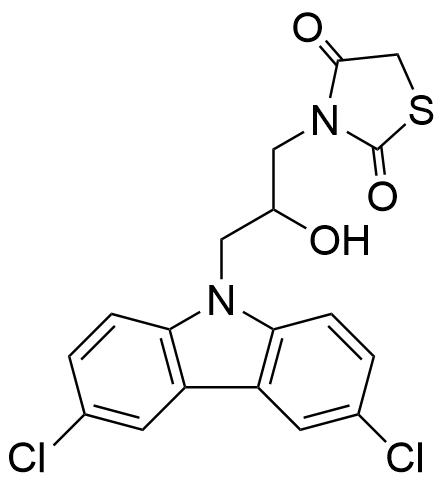 10074-A4[415-416] | MYC; MYC-MAX | 抑制MYC二聚体形成 |
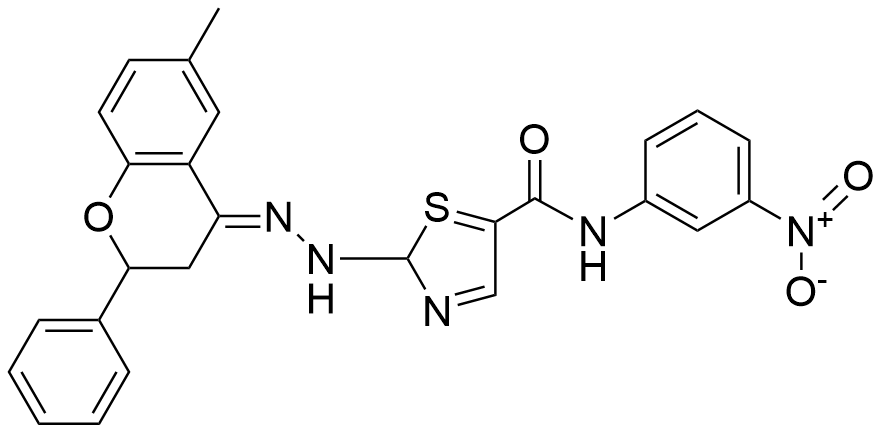 10050-C10[415] | MYC; MYC-MAX | 抑制MYC二聚体形成 |
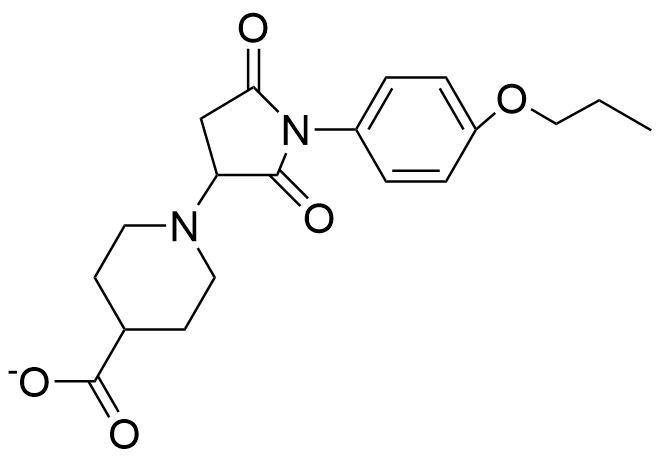 10031-B8[415-416] | MYC; MYC-MAX | 抑制MYC二聚体形成 |
 10075-G5[415-416] | MYC; MYC-MAX | 抑制MYC二聚体形成 |
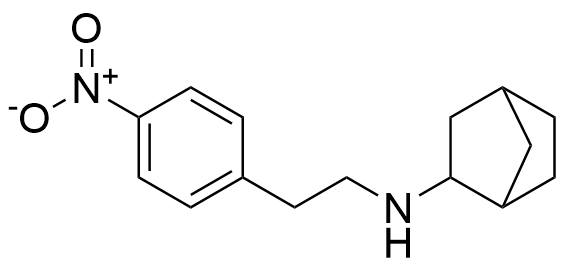 10009-G9[415-416] | MYC; MYC-MAX | 抑制MYC二聚体形成 |
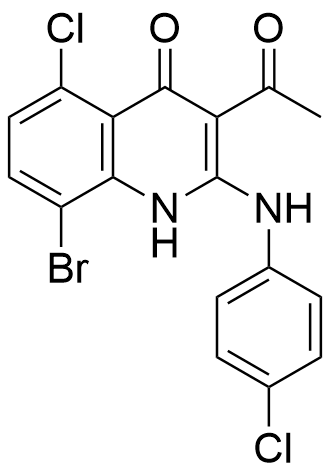 KSI-3716[426-427] | MYC-MAX-DNA | 抑制蛋白质-DNA相互作用 |
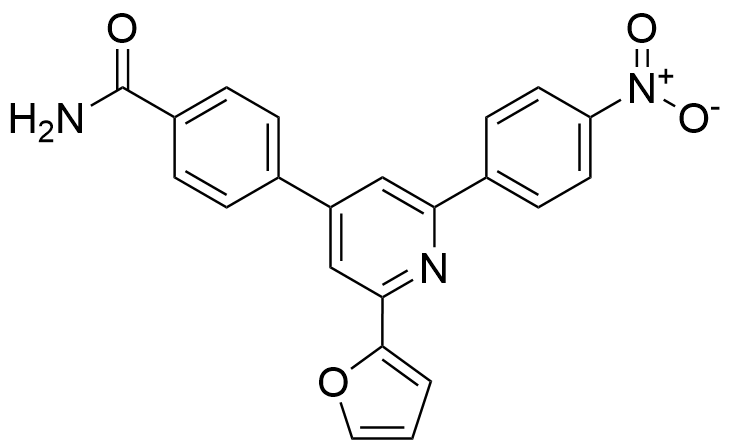 KJ-Pyr-9[428] | MYC; MYC-MAX | 抑制MYC二聚体形成 |
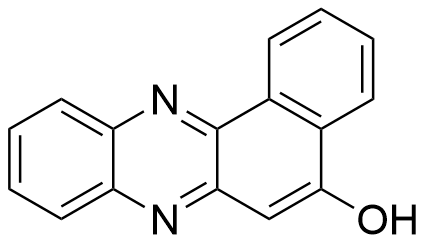 sAJM589[429] | MYC-MAX | 抑制MYC二聚体形成 |
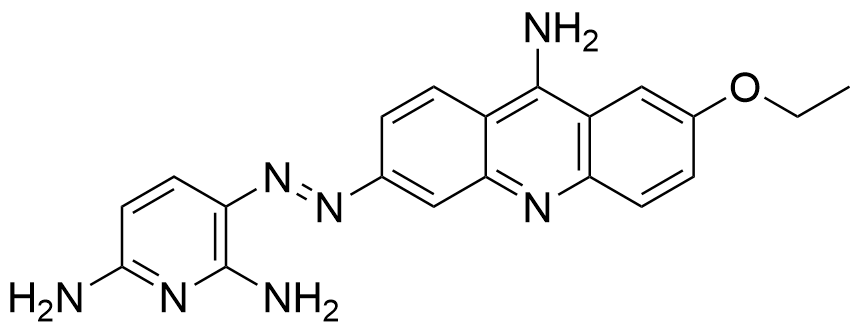 MYCMI-6[430] | MYC | 抑制MYC二聚体形成 |
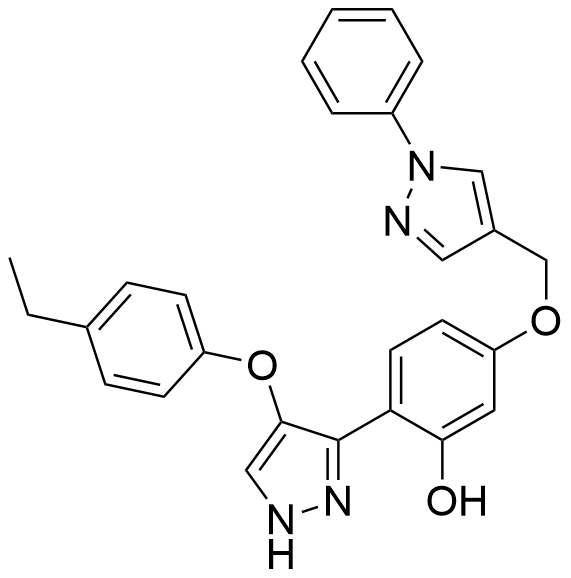 Min9[433] | MYC | 抑制MYC二聚体形成 |
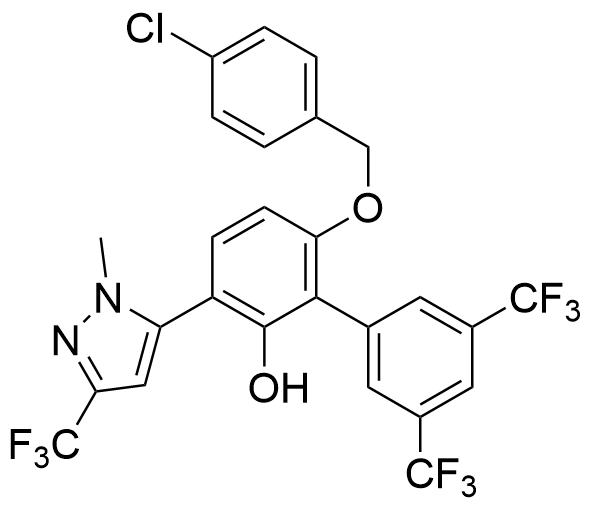 MYCi361[433] | MYC | 抑制MYC二聚体形成 |
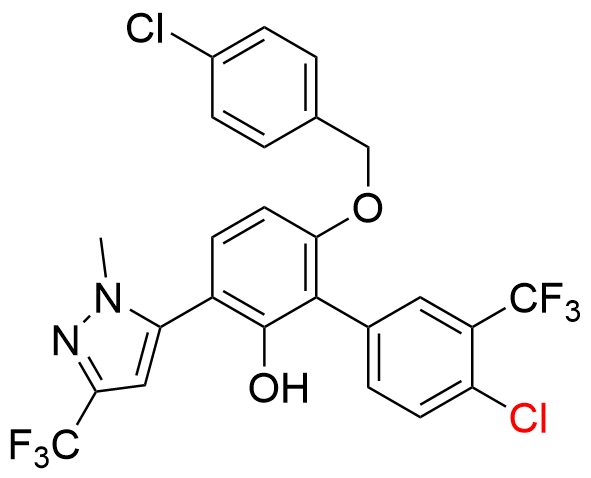 MYCi975[433] | MYC | 抑制MYC二聚体形成 |
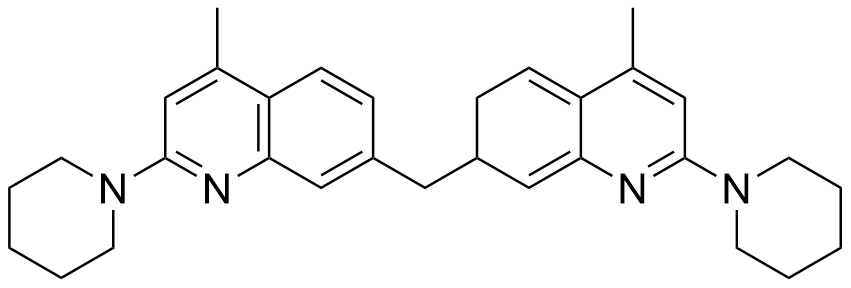 NSC13728[434,437] | MAX-MAX | 稳定MAX-MAX, 降低MYC蛋白水平 |
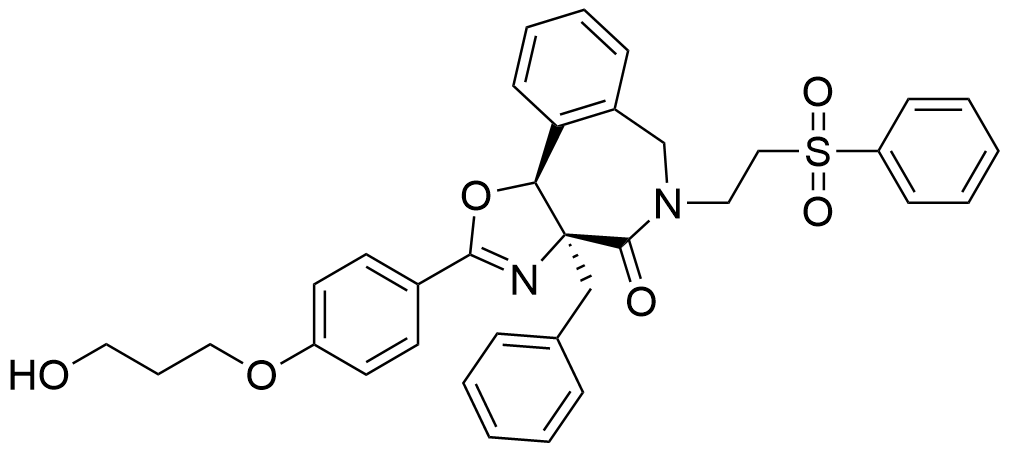 KI-MS2-001[436] | MAX-MAX | 稳定MAX-MAX, 降低MYC蛋白水平 |
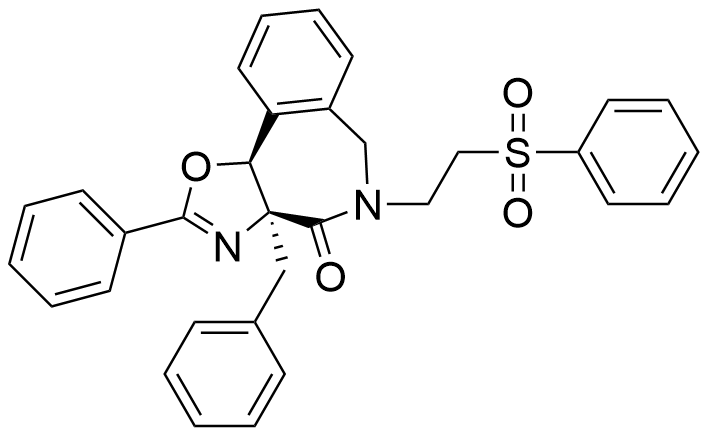 KI-MS2-008[436] | MAX-MAX | 稳定MAX-MAX, 降低MYC蛋白水平 |