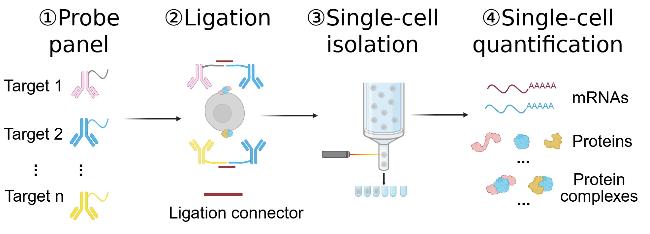
1 引言
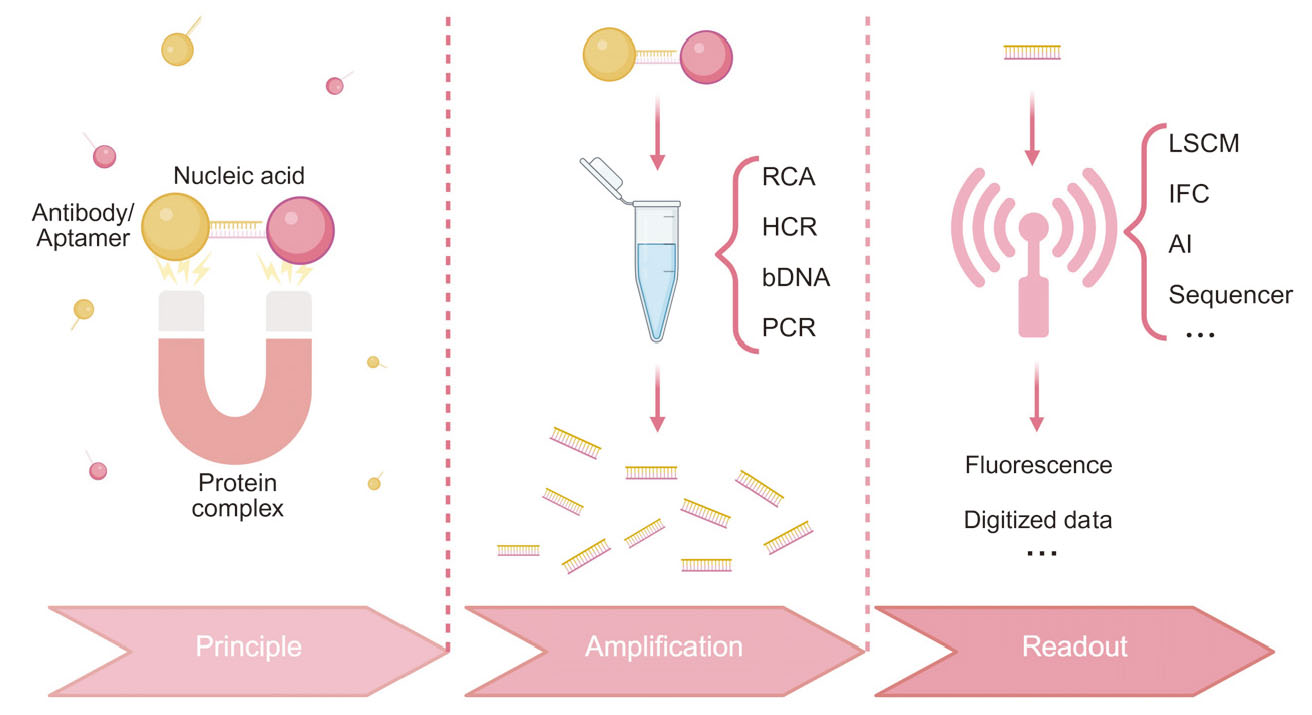
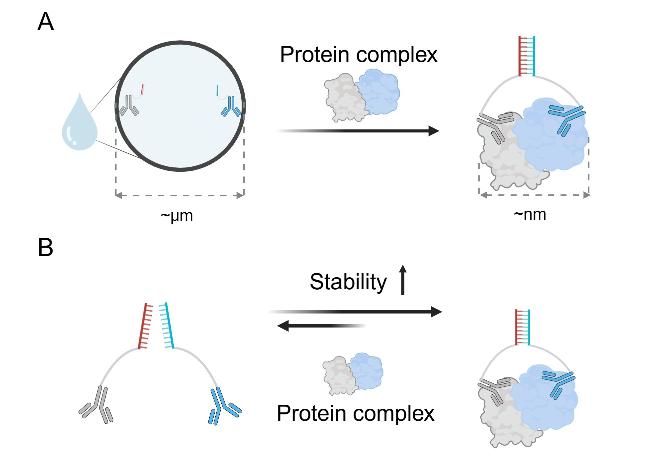
2 邻近依赖性核酸自组装用于PPI检测的基本原理
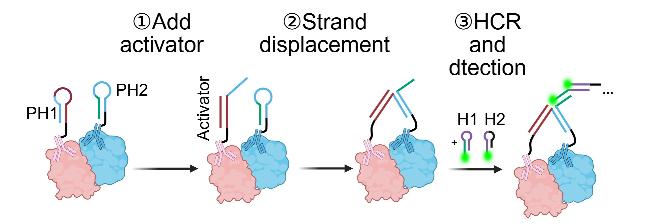
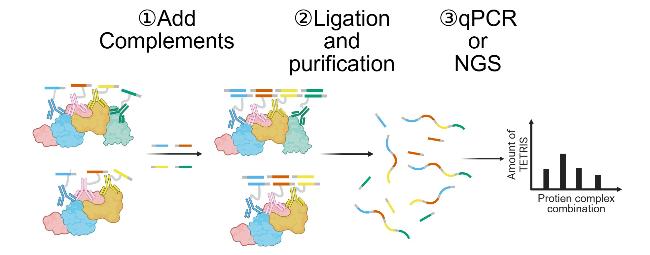
3 信号放大技术
表1 不同信号放大方法用于PPI检测的比较Table 1 Comparison of different signal amplification methods for PPI detection |
| 信号放大方法 | RCA | HCR | bDNA | PCR |
|---|---|---|---|---|
| 核心机制 | 酶促等温扩增 | 非酶促等温自组装 | 非酶促多级探针杂交 | 酶促热循环指数扩增 |
| 成本 | 中 | 低 | 中 | 高 |
| 商品化试剂盒 | 有 | 有 | 无 | 无 |
| 操作复杂度 | 中 依赖两次酶促反应, 相 对简单 | 低 无酶, 仅需1~2轮孵育 和洗涤, 反应条件简单 | 高 无酶, 但需要3~4轮孵育和 洗涤, 对操作者的要求较高 | 中 依赖至少两次酶促反应 及专用热循环仪 |
| 误差来源 | (1) DNA连接酶活性波动 (2) Phi29聚合酶活性波动 (3)高丰度时的空间信号重叠 | (1)非特异性发夹自组装 (2)反应环境中盐离子浓 度等影响组装效率 | (1)多轮孵育、洗涤操作导致 误差 (2)探针的非特异性杂交 | (1) DNA连接酶活性波动 (2) PCR的序列偏好性、 保真度 |