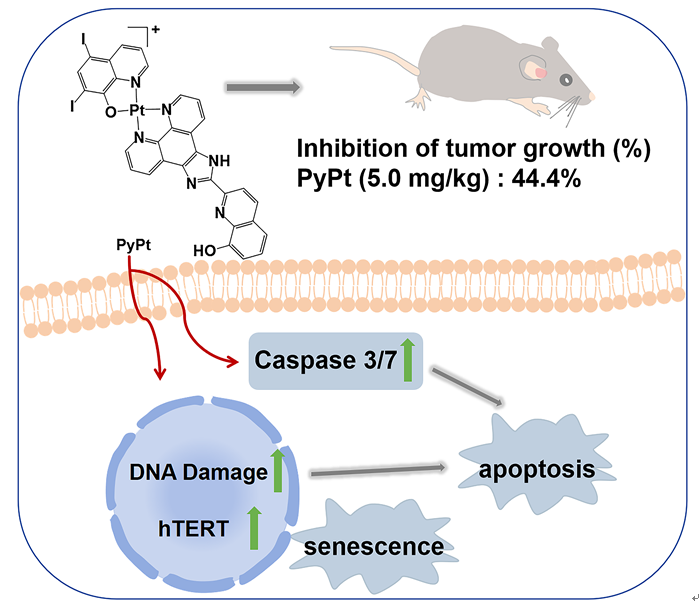
1 引言
2 结果与讨论
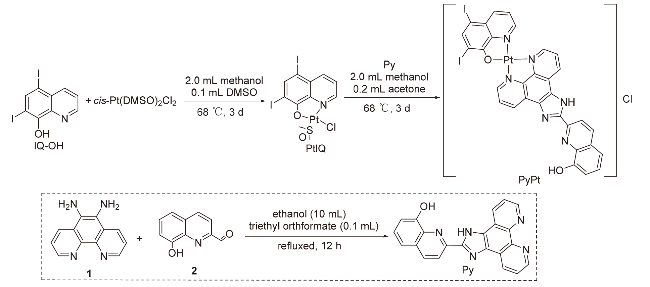
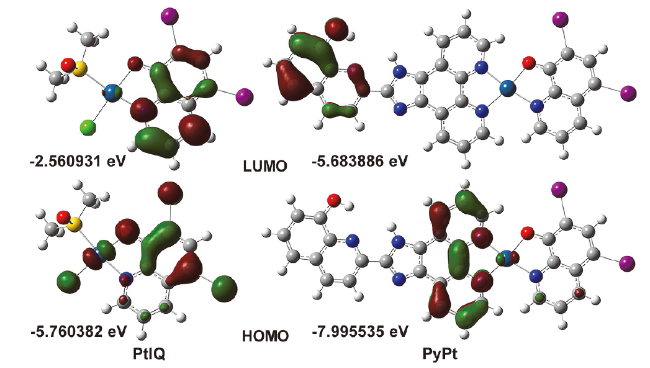
2.1 PyPt和PtIQ的合成与热力学稳定性研究
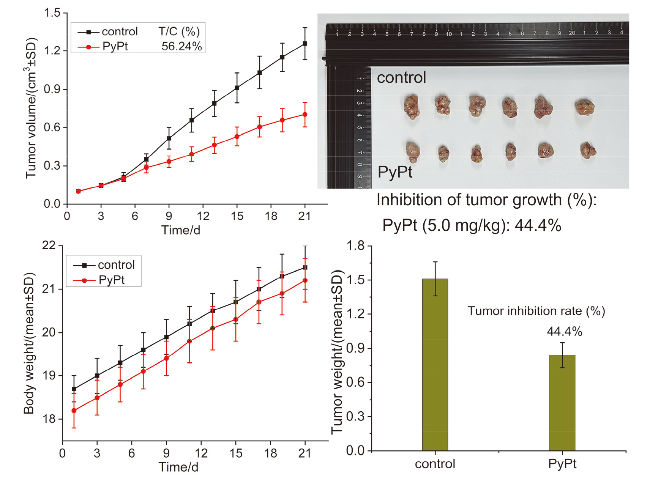
2.2 抗癌活性研究
表1 顺铂、PyPt和PtIQ对MDA-MB-231和HL-7702细胞的IC50值(μmol/L)Table 1 The IC50 values of cisplatin, PyPt and PtIQ against MDA-MB-231 and HL-7702 cells |
| Entrya | A549 | A549/DDP | MDA-MB- 231 | HL-7702 | SIb |
|---|---|---|---|---|---|
| Py | >50.0 | >50.0 | >50.0 | >50.0 | ca. 1.0 |
| IQ-OH | >50.0 | >50.0 | >50.0 | >50.0 | ca. 1.0 |
| cis-[PtCl2- (DMSO)2] | >50.0 | >50.0 | >50.0 | >50.0 | ca. 1.0 |
| PtIQ | 7.2±0.5 | 6.1±0.9 | 9.0±0.1 | 35.5±0.4 | 4.0 |
| PyPt | 1.0±0.2 | 0.9±0.3 | 0.05±0.04 | >50.0 | >980.4 |
| PtIQ+Py | 7.0±0.5 | 6.0±0.1 | 8.8±0.4 | 36.2±0.7 | 4.2 |
| 顺铂 | 13.0±0.6 | >50.0 | 11.1±0.3 | 15.6±0.2 | 1.4 |
a The cells were incubated for 48 h; b Tumor selectivity (SI)=IC50 value (HL-7702)/IC50 value (MDA-MB-231). |
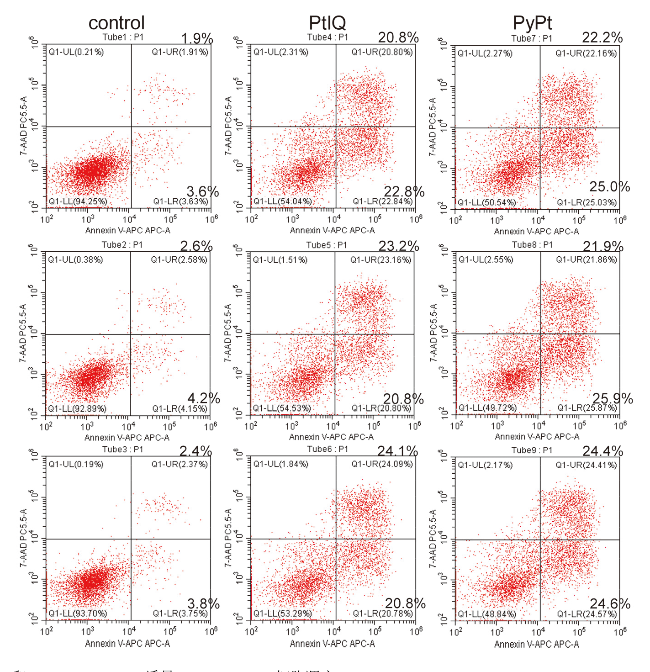
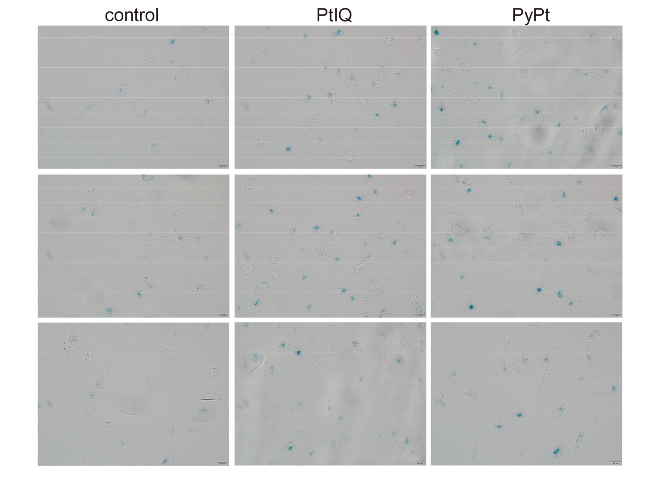
2.3 PyPt和PtIQ诱导细胞凋亡和衰老
图3 PyPt(0.05 μmol/L)和PtIQ (9.0 μmol/L)诱导MDA-MB-231细胞凋亡Figure 3 Apoptosis of MDA-MB-231 cells induced by PyPt (0.05 μmol/L) and PtIQ (9.0 μmol/L) |
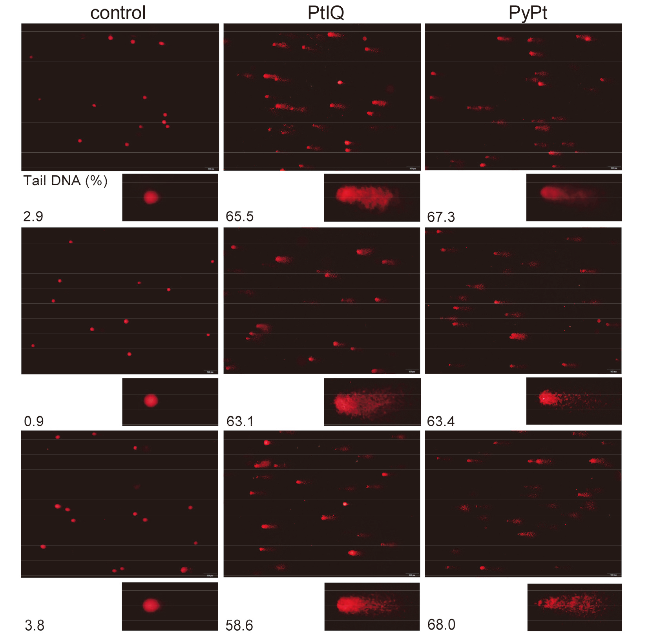
2.4 DNA损伤实验
图5 采用彗星试验检测PyPt (0.05 μmol/L)和PtIQ (9.0 μmol/L)作用MDA-MB-231细胞48 h后DNA损伤的情况Figure 5 Induction of DNA damage in MDA-MB-231 cells by PyPt (0.05 μmol/L) and PtIQ (9.0 μmol/L) for 48 h using neutral comet assay Image multiple: 100 μm. |
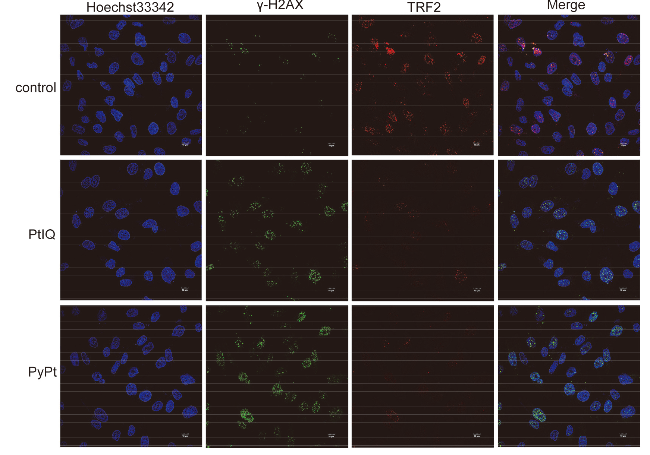
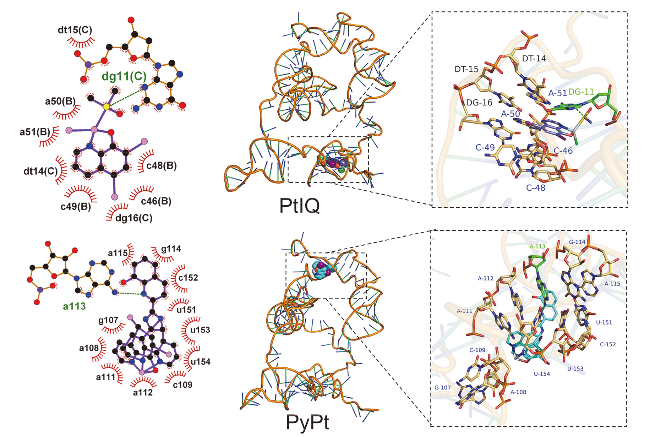
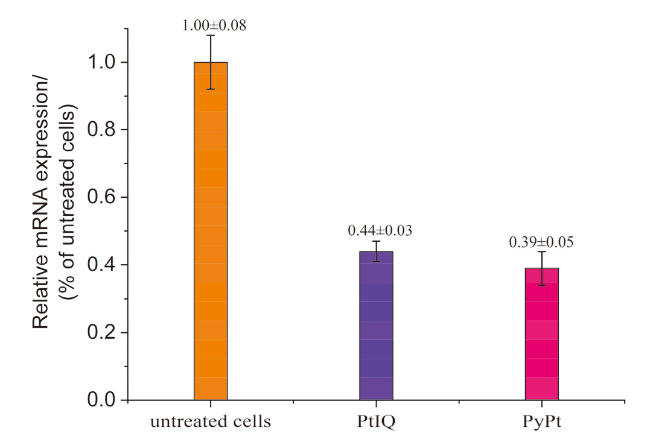
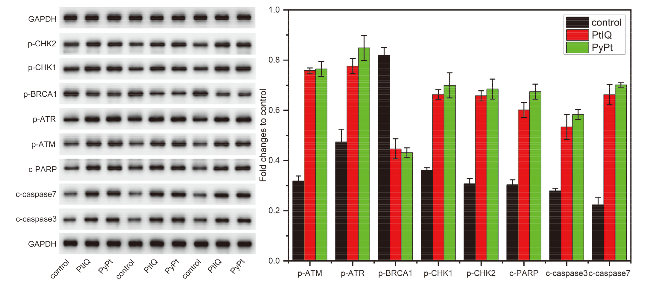
2.5 分子对接与RT-qPCR实验
图7 分子对接研究PyPt和PtIQ与目标蛋白hTERT中DNAFigure 7 Molecular docking analysis of PyPt and PtIQ against the target hTERT DNA |