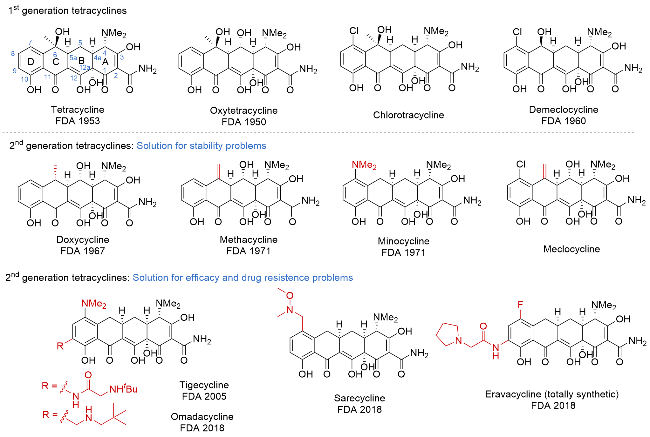
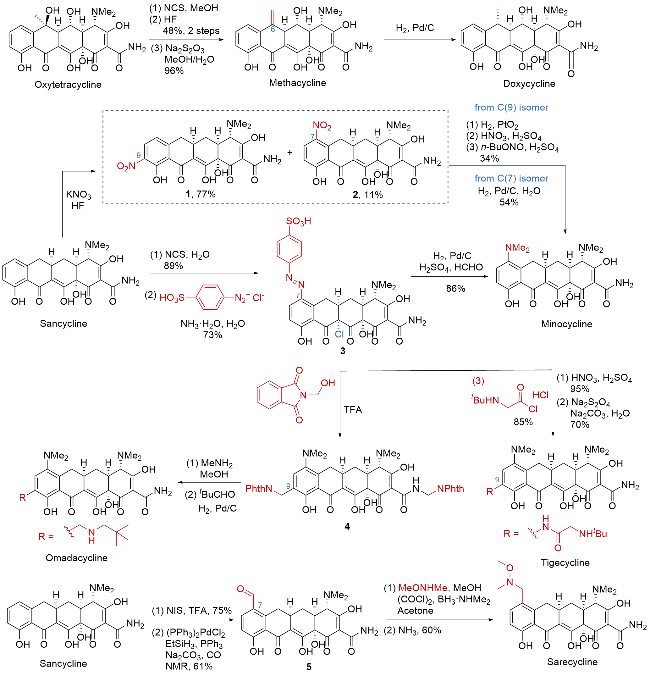
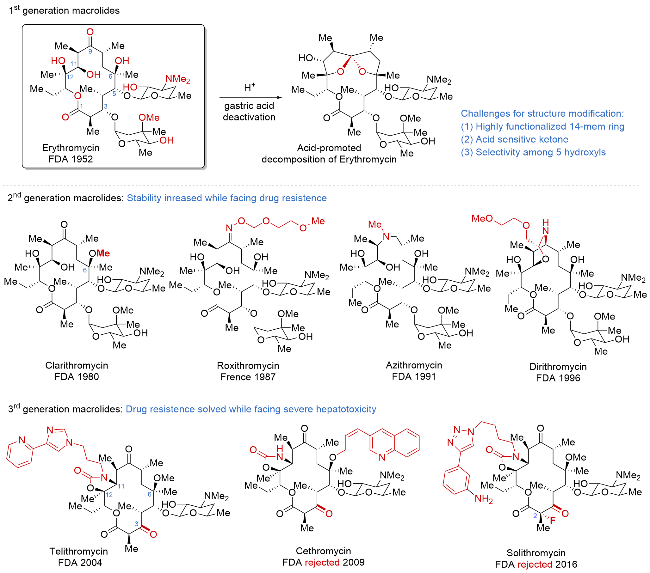
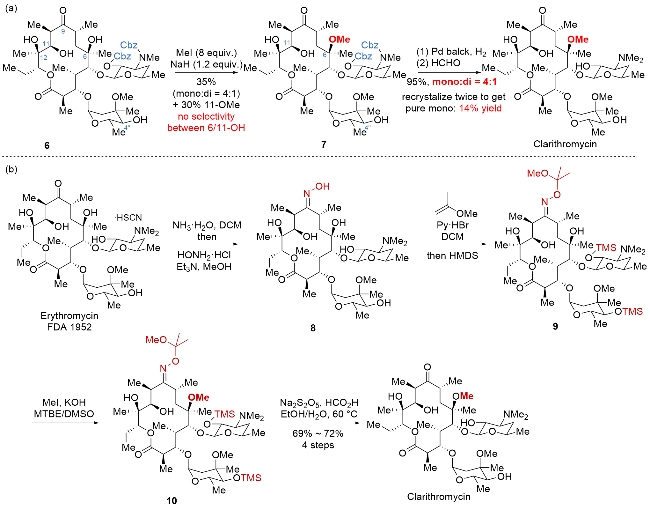
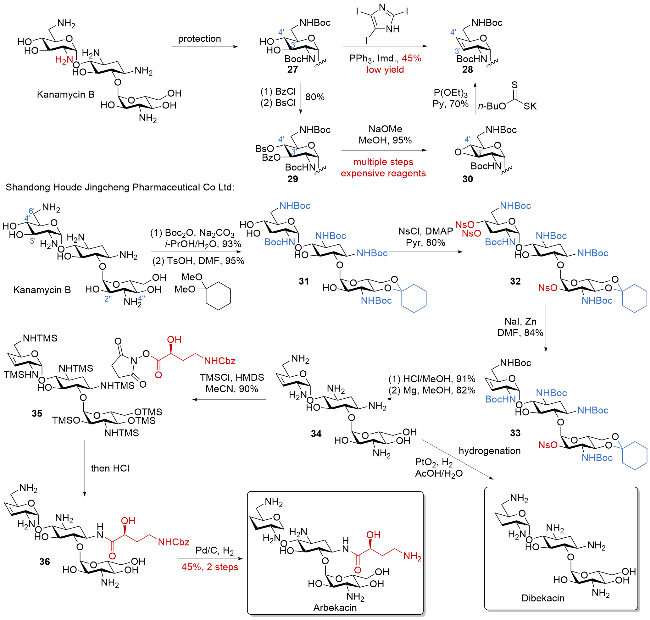
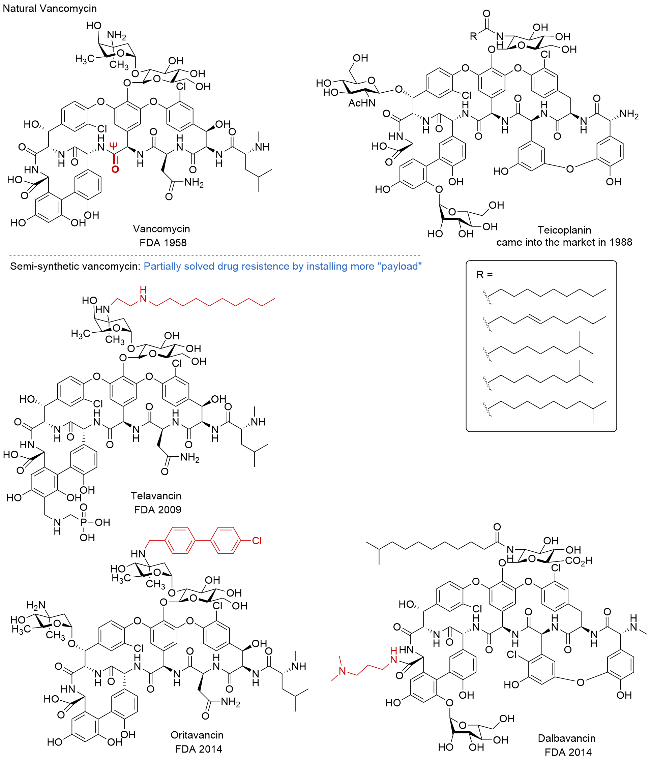
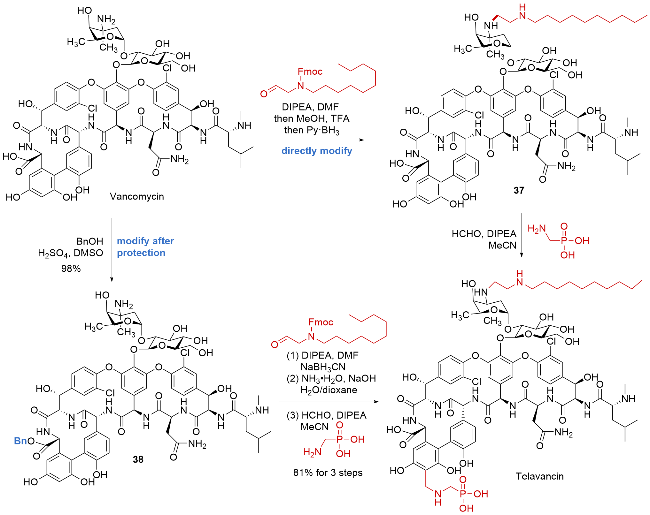
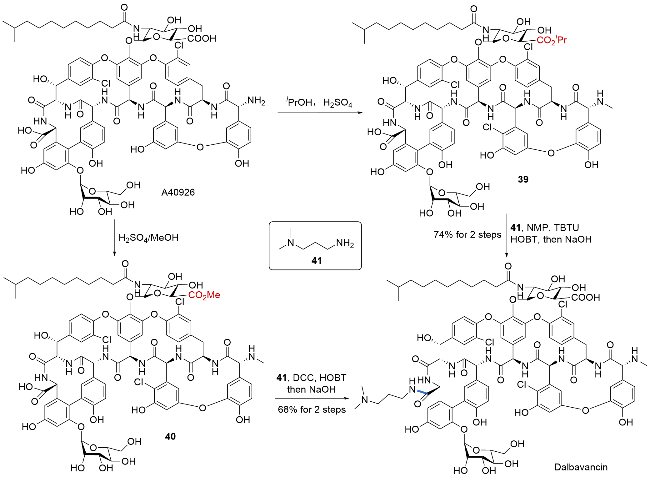
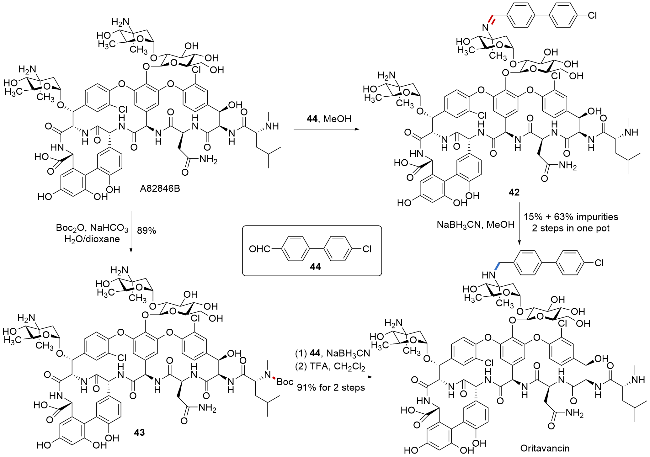
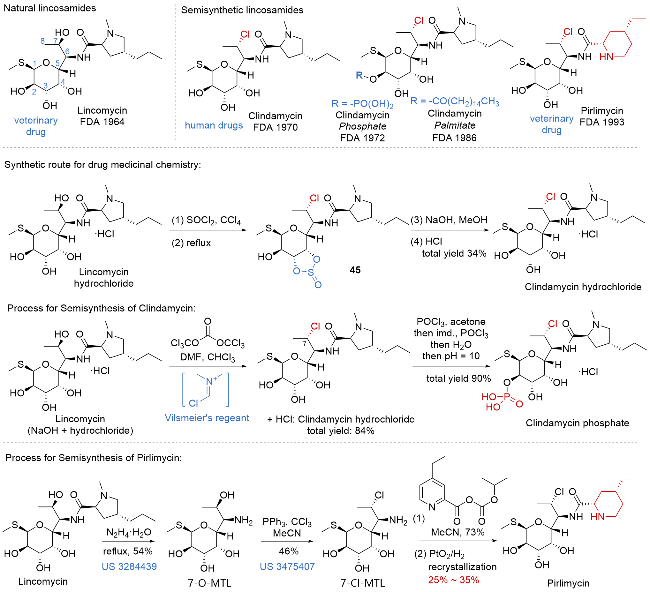
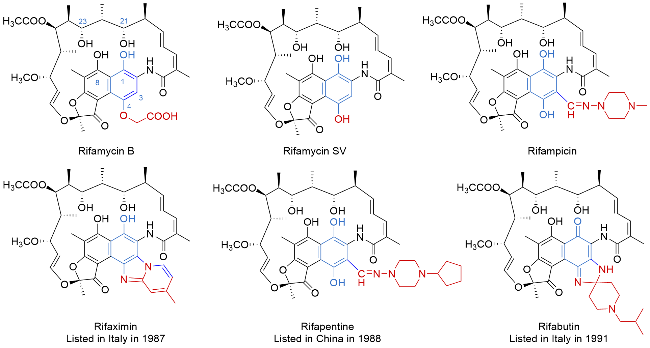
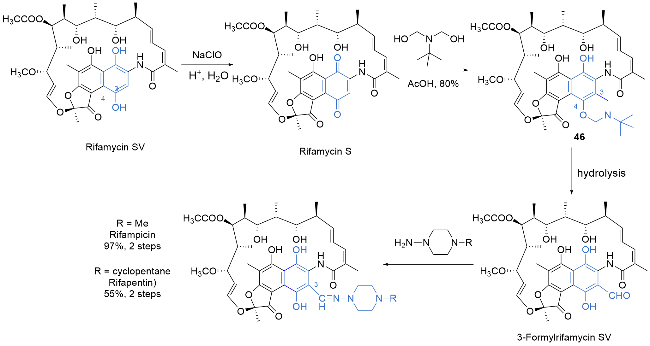
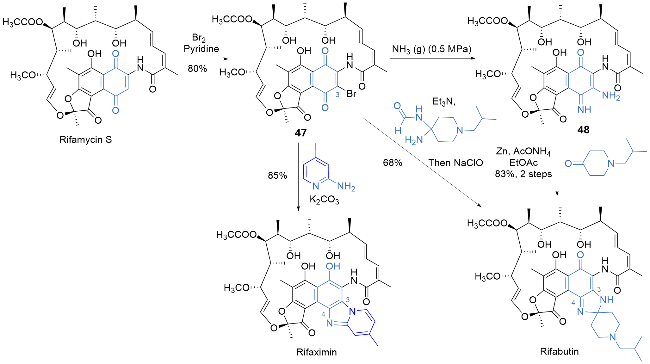
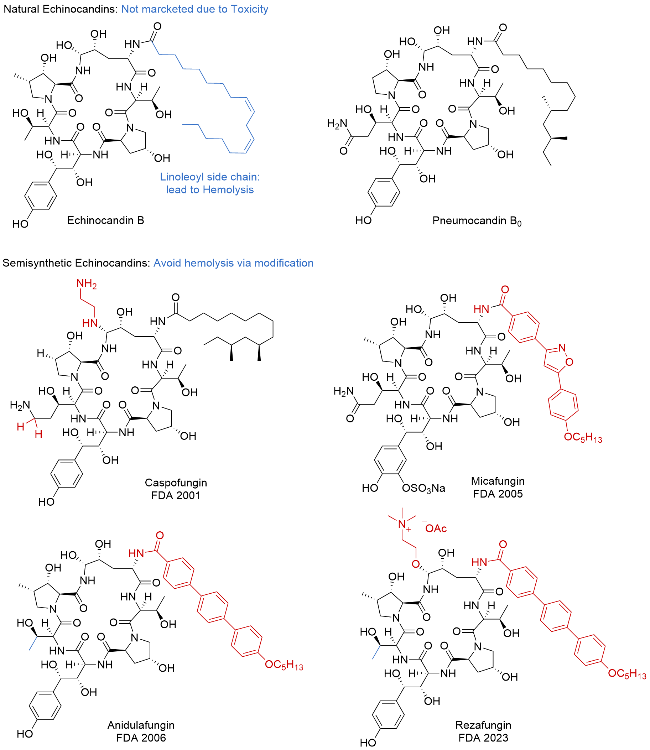
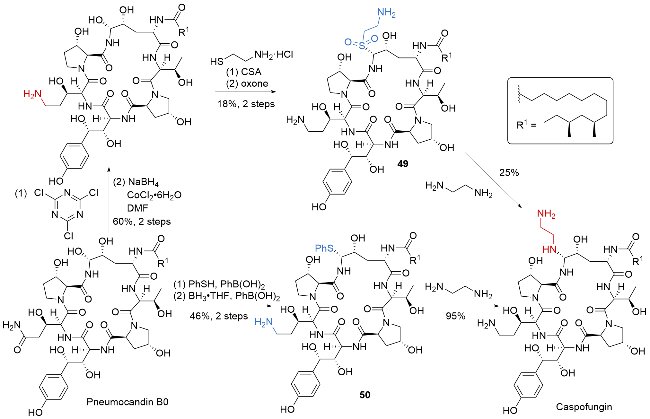
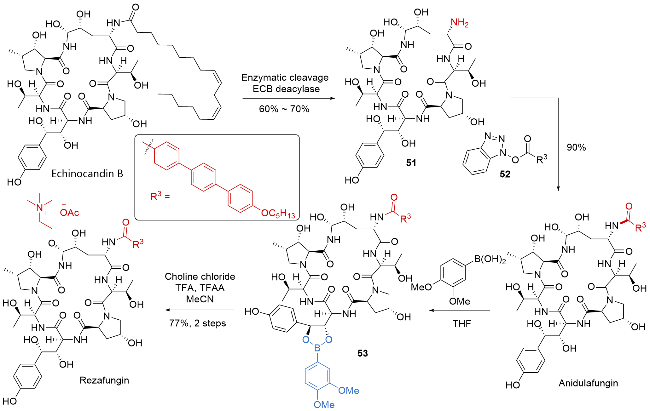
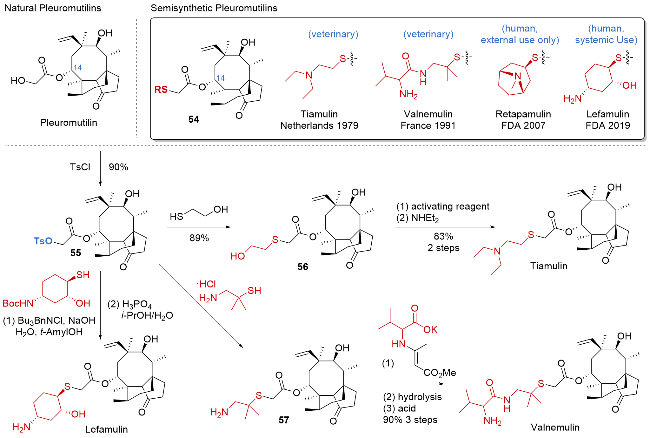
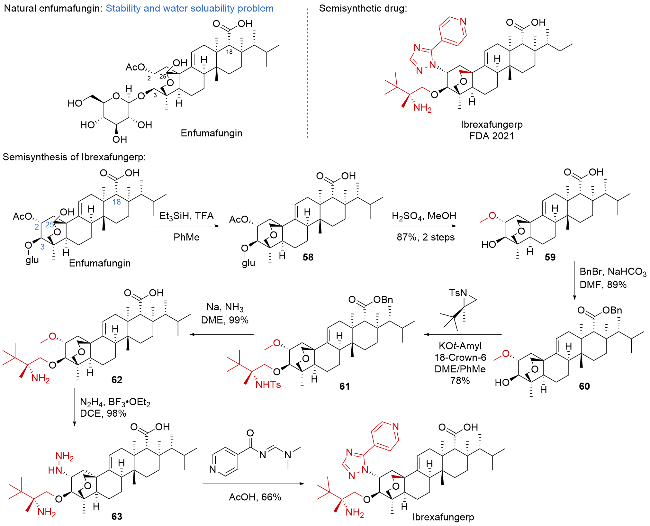
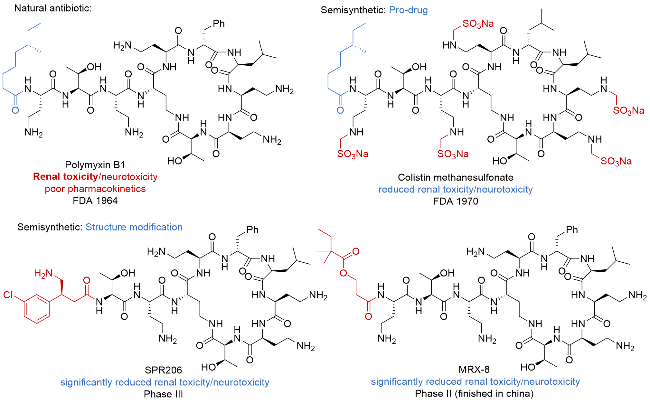
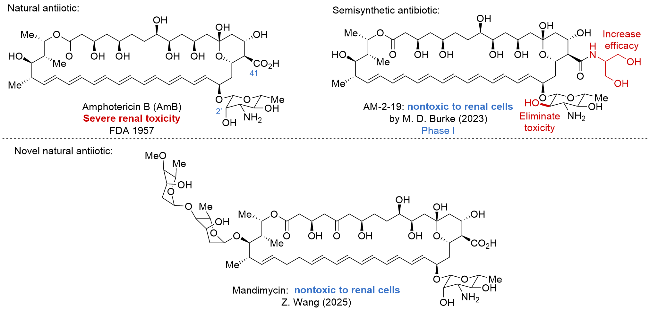
| [32] |
(a) Quirke, J. C. K.; Rajasekaran, P.; Sarpe, V. A.; Sonousi, A.; Osinnii, I.; Gysin, M.; Haldimann, K.; Fang, Q.-J.; Shcherbakov, D.; Hobbie, S. N.; Sha, S.-H.; Schacht, J.; Vasella, A.; Böttger, E. C.; Crich, D. J. Am. Chem. Soc. 2020, 142, 530.
(b) Matsushita, T.; Sati, G. C.; Kondasinghe, N.; Pirrone, M. G.; Kato, T.; Waduge, P.; Kumar, H. S.; Sanchon, A. C.; Dobosz- Bartoszek, M.; Shcherbakov, D.; Juhas, M.; Hobbie, S. N.; Schrepfer, T.; Chow, C. S.; Polikanov, Y. S.; Schacht, J.; Vasella, A.; Böttger, E. C.; Crich, D. J. Am. Chem. Soc. 2019, 141, 5051.
DOI
PMID
(c) Mandhapati, A. R.; Yang, G.; Kato, T.; Shcherbakov, D.; Hobbie, S. N.; Vasella, A.; Böttger, E. C.; Crich, D. J. Am. Chem. Soc. 2017, 139, 14611.
DOI
PMID
(d) Matsushita, T.; Chen, W.; Juskeviciene, R.; Teo, Y.; Shcherbakov, D.; Vasella, A.; Böttger, E. C.; Crich, D. J. Am. Chem. Soc. 2015, 137, 7706.
DOI
PMID
(e) Lubriks, D.; Zogota, R.; Sarpe, V. A.; Matsushita, T.; Sati, G. C.; Haldimann, K.; Gysin, M.; Böttger, E. C.; Vasella, A.; Suna, E.; Hobbie, S. N.; Crich, D. ACS Infect. Dis. 2021, 7, 2413.
|