有机化学 ›› 2021, Vol. 41 ›› Issue (11): 4279-4288.DOI: 10.6023/cjoc202105035 上一篇 下一篇
综述与进展
乔怡a, 张庆林b,c, 陈单丹b,c,*( ), 刘美娜a,*(
), 刘美娜a,*( ), 刘文b,c,*(
), 刘文b,c,*( )
)
收稿日期:2021-05-20
修回日期:2021-06-16
发布日期:2021-07-05
通讯作者:
陈单丹, 刘美娜, 刘文
基金资助:
Yi Qiaoa, Qinglin Zhangb,c, Dandan Chenb,c( ), Meina Liua(
), Meina Liua( ), Wen Liub,c(
), Wen Liub,c( )
)
Received:2021-05-20
Revised:2021-06-16
Published:2021-07-05
Contact:
Dandan Chen, Meina Liu, Wen Liu
Supported by:文章分享

作为临床药物的重要来源, 放线菌天然产物及其衍生物已被大规模开发并应用于临床. 为了实现放线菌来源活性天然产物的高效合成、新型天然产物的结构衍生以及未知天然产物的定向挖掘, 往往需要聚焦于生物合成基因簇, 运用生物学技术开展理性的遗传改造. 规律性成簇间隔的短回文重复序列及相关蛋白(CRISPR/Cas)是部分细菌和古细菌在长期自然进化过程中发展而来的一种对抗外源遗传物质入侵的适应性免疫防御系统. 以CRISPR/Cas9为代表的基因编辑系统已被广泛应用于脱氧核糖核酸(DNA)片段的靶向特异性切割, 为生物技术领域的发展提供了无限可能. 综述了CRISPR/Cas9系统在放线菌遗传改造中的应用情况, 包括在基因簇抓取、编辑与重构、原位同源重组以及代谢调控方面的研究进展, 涉及全新的CRISPR/Cas9介导的基因编辑方法以及代表性的成功案例, 为高效利用CRISPR/Cas9系统获取放线菌天然产物提供了借鉴.
乔怡, 张庆林, 陈单丹, 刘美娜, 刘文. CRISPR/Cas9基因编辑系统在获取放线菌天然产物中的应用[J]. 有机化学, 2021, 41(11): 4279-4288.
Yi Qiao, Qinglin Zhang, Dandan Chen, Meina Liu, Wen Liu. Application of CRISPR/Cas9 Gene Editing System in Obtaining Natural Products in Actinomycetes[J]. Chinese Journal of Organic Chemistry, 2021, 41(11): 4279-4288.
| 方法名称 | 核心技术 | 操作体系 | 技术参数 | 方法特点 |
|---|---|---|---|---|
| CRISPR/Cas9介导的TAR克隆 | CRISPR/Cas9 TAR | 大肠杆菌 酿酒酵母 | 抓取上限约60~70 kb | 操作体系复杂、抓取片段较短 |
| CATCH | CRISPR/Cas9 Gibson组装 | 体外 大肠杆菌 | 抓取上限超过100 kb | 技术要求较高、片段越长抓取越难 |
| ExoCET | CRISPR/Cas9 RecET重组 | 大肠杆菌 | 抓取上限超过100 kb | 操作体系相对简单、抓取片段较长 |
| ICE | CRISPR/Cas9 T4聚合酶修复 Gibson组装 | 体外 大肠杆菌 | 单位点编辑 | 大片段编辑的技术要求较高 |
| mCRISTAR | CRISPR/Cas9 TAR | 大肠杆菌 酿酒酵母 | 2~4个位点同时编辑 | 操作较为简便 |
| mpCRISTAR | CRISPR/Cas9 TAR | 大肠杆菌 酿酒酵母 | 4~8个位点同时编辑 | 操作较为灵活、步骤较多 |
| miCASTAR | CRISPR/Cas9 TAR | 体外 酿酒酵母 | 2~4个位点同时编辑 | 操作体系相对简单、操作简便 |
| 方法名称 | 核心技术 | 操作体系 | 技术参数 | 方法特点 |
|---|---|---|---|---|
| CRISPR/Cas9介导的TAR克隆 | CRISPR/Cas9 TAR | 大肠杆菌 酿酒酵母 | 抓取上限约60~70 kb | 操作体系复杂、抓取片段较短 |
| CATCH | CRISPR/Cas9 Gibson组装 | 体外 大肠杆菌 | 抓取上限超过100 kb | 技术要求较高、片段越长抓取越难 |
| ExoCET | CRISPR/Cas9 RecET重组 | 大肠杆菌 | 抓取上限超过100 kb | 操作体系相对简单、抓取片段较长 |
| ICE | CRISPR/Cas9 T4聚合酶修复 Gibson组装 | 体外 大肠杆菌 | 单位点编辑 | 大片段编辑的技术要求较高 |
| mCRISTAR | CRISPR/Cas9 TAR | 大肠杆菌 酿酒酵母 | 2~4个位点同时编辑 | 操作较为简便 |
| mpCRISTAR | CRISPR/Cas9 TAR | 大肠杆菌 酿酒酵母 | 4~8个位点同时编辑 | 操作较为灵活、步骤较多 |
| miCASTAR | CRISPR/Cas9 TAR | 体外 酿酒酵母 | 2~4个位点同时编辑 | 操作体系相对简单、操作简便 |
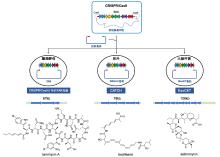

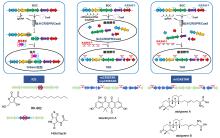

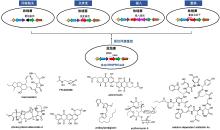

| 质粒名称 | 抗性标签 | cas9基因启动子 | sgRNA启动子 | 质粒特性 |
|---|---|---|---|---|
| pCRISPomyces-1 | 阿伯拉霉素 | 组成型rpsLp | 组成型gapdhp (crRNA) 组成型rpsLp (tracrRNA) | 来源于pSG5的温敏型复制子 |
| pCRISPomyces-2 | 阿伯拉霉素 | 组成型rpsLp | 组成型gapdhp (sgRNA) | 来源于pSG5的温敏型复制子 |
| pKCcas9dO | 阿伯拉霉素 | 诱导型tipAp | 组成型j23199p (sgRNA) | 来源于pSG5的温敏型复制子 |
| pCRISPR-Cas9 | 阿伯拉霉素 硫链丝菌素 | 诱导型tipAp | 组成型ermE*p (sgRNA) | 来源于pSG5的温敏型复制子 |
| pCRISPR-dCas9 | 阿伯拉霉素 硫链丝菌素 | 诱导型tipAp | 组成型ermE*p (sgRNA) | 来源于pSG5的温敏型复制子 |
| pWHU2653 | 阿伯拉霉素 | 组成型aac(3) IVp | 组成型ermE*p (sgRNA) | 来源于pIJ101的复制子 CodA 反筛标签 |
| pMWCas9 | 阿伯拉霉素 硫链丝菌素 | 诱导型tipAp | 组成型ermE*p (sgRNA) | 来源于pIJ101的复制子 CodA反筛标签 |
| 质粒名称 | 抗性标签 | cas9基因启动子 | sgRNA启动子 | 质粒特性 |
|---|---|---|---|---|
| pCRISPomyces-1 | 阿伯拉霉素 | 组成型rpsLp | 组成型gapdhp (crRNA) 组成型rpsLp (tracrRNA) | 来源于pSG5的温敏型复制子 |
| pCRISPomyces-2 | 阿伯拉霉素 | 组成型rpsLp | 组成型gapdhp (sgRNA) | 来源于pSG5的温敏型复制子 |
| pKCcas9dO | 阿伯拉霉素 | 诱导型tipAp | 组成型j23199p (sgRNA) | 来源于pSG5的温敏型复制子 |
| pCRISPR-Cas9 | 阿伯拉霉素 硫链丝菌素 | 诱导型tipAp | 组成型ermE*p (sgRNA) | 来源于pSG5的温敏型复制子 |
| pCRISPR-dCas9 | 阿伯拉霉素 硫链丝菌素 | 诱导型tipAp | 组成型ermE*p (sgRNA) | 来源于pSG5的温敏型复制子 |
| pWHU2653 | 阿伯拉霉素 | 组成型aac(3) IVp | 组成型ermE*p (sgRNA) | 来源于pIJ101的复制子 CodA 反筛标签 |
| pMWCas9 | 阿伯拉霉素 硫链丝菌素 | 诱导型tipAp | 组成型ermE*p (sgRNA) | 来源于pIJ101的复制子 CodA反筛标签 |
| [1] |
Bérdy, J. J. Antibiot. (Tokyo) 2012, 65, 441.
doi: 10.1038/ja.2012.54 |
| [2] |
Newman, D. J.; Cragg, G. M. J. Nat. Prod. 2020, 83, 770.
doi: 10.1021/acs.jnatprod.9b01285 pmid: 32162523 |
| [3] |
Bode, H. B.; Müller, R. Angew. Chem., Int. Ed. 2005, 44, 6828.
doi: 10.1002/(ISSN)1521-3773 |
| [4] |
Lin, Z.; Chen, D.; Liu, W. Sci. China Chem. 2016, 59, 1175.
doi: 10.1007/s11426-016-0062-x |
| [5] |
Chen, M.; Liu, J.; Duan, P. Natl. Sci. Rev. 2017, 4, 553.
doi: 10.1093/nsr/nww045 |
| [6] |
Smanski, M. J.; Zhou, H.; Claesen, J. Nat. Rev. Microbiol. 2016, 14, 135.
doi: 10.1038/nrmicro.2015.24 |
| [7] |
Baltz, R. H. J. Ind. Microbiol. Biotechnol. 2016, 43, 343.
doi: 10.1007/s10295-015-1682-x |
| [8] |
Medema, M. H.; Blin, K.; Cimermancic, P. Nucleic Acids Res. 2011, 39, W339.
doi: 10.1093/nar/gkr466 |
| [9] |
Ziemert, N.; Alanjary, M.; Weber, T. Nat. Prod. Rep. 2016, 33, 988.
doi: 10.1039/c6np00025h pmid: 27272205 |
| [10] |
Horvath, P.; Barrangou, R. Science 2010, 327, 167.
doi: 10.1126/science.1179555 |
| [11] |
Smargon, A. A.; Shi, Y. J.; Yeo, G. W. Nat. Cell Biol. 2020, 22, 143.
doi: 10.1038/s41556-019-0454-7 pmid: 32015437 |
| [12] |
Cong, L.; Ran, F. A.; Cox, D. Science 2013, 339, 819.
doi: 10.1126/science.1231143 pmid: 23287718 |
| [13] |
Gasiunas, G.; Barrangou, R.; Horvath, P. Proc. Natl. Acad. Sci. U. S. A. 2012, 109, E2579.
doi: 10.1073/pnas.1208507109 |
| [14] |
Fonfara, I.; Le, R. A.; Chylinski, K. Nucleic Acids Res. 2014, 42, 2577.
doi: 10.1093/nar/gkt1074 pmid: 24270795 |
| [15] |
Hsu, P. D.; Lander, E. S.; Zhang, F. Cell 2014, 157, 1262.
doi: 10.1016/j.cell.2014.05.010 |
| [16] |
Jinek, M.; Chylinski, K.; Fonfara, I.; Hauer, M.; Doudna, J. A.; Charpentier, E. Science 2012, 337, 816.
doi: 10.1126/science.1225829 |
| [17] |
Blin, K.; Pedersen, L. E.; Weber, T. Synth. Syst. Biotechnol. 2016, 1, 118.
doi: 10.1016/j.synbio.2016.01.003 pmid: 29062934 |
| [18] |
Burke, D. T.; Carle, G. F.; Olson, M. V. Biotechnol. 1992, 24, 172.
|
| [19] |
Fu, J.; Bian, X.; Hu, S. Nat. Biotechnol. 2012, 30, 440.
doi: 10.1038/nbt.2183 |
| [20] |
Noskov, V.; Kouprina, N.; Leem, S. H. Nucleic Acids Res. 2002, 30, e8.
doi: 10.1093/nar/30.2.e8 |
| [21] |
Hoang, T. T.; Karkhoff-Schweizer, R. R.; Kutchma, A. J. Gene 1998, 212, 77.
pmid: 9661666 |
| [22] |
Li, L.; Jiang, W.; Lu, Y. Biotechnol. Adv. 2017, 35, 936.
doi: 10.1016/j.biotechadv.2017.03.007 |
| [23] |
Yamanaka, K.; Reynolds, K. A.; Kersten, R. D. Proc. Natl. Acad. Sci. U. S. A. 2014, 111, 1957.
doi: 10.1073/pnas.1319584111 pmid: 24449899 |
| [24] |
Lee, N. C.; Larionov, V.; Kouprina, N. Nucleic Acids Res. 2015, 43, e55.
doi: 10.1093/nar/gkv112 |
| [25] |
Jiang, W.; Zhao, X.; Gabrieli, T.; Lou, C. B.; Ebenstein, Y.; Zhu, T. F. Nat. Commun. 2015, 6, 8101.
doi: 10.1038/ncomms9101 |
| [26] |
Jiang, W.; Zhu, T. F. Nat. Protoc. 2016, 11, 960.
doi: 10.1038/nprot.2016.055 |
| [27] |
Wang, H.; Li, Z.; Jia, R. Nucleic Acids Res. 2018, 46, 2697.
doi: 10.1093/nar/gkx1296 |
| [28] |
Wang, J. W.; Wang, A.; Li, K. BioTechniques 2015, 58, 161.
doi: 10.2144/000114261 |
| [29] |
Kang, H. S.; Charlop-Powers, Z.; Brady, S. F. ACS Synth. Biol. 2016, 5, 1002.
doi: 10.1021/acssynbio.6b00080 |
| [30] |
Kim, H.; Ji, C.-H.; Je, H.-W. ACS Synth. Biol. 2020, 9, 175.
doi: 10.1021/acssynbio.9b00382 |
| [31] |
Kim, S. H.; Lu, W.; Ahmadi, M. K. ACS Synth. Biol. 2019, 8, 109.
doi: 10.1021/acssynbio.8b00361 |
| [32] |
Jiang, Y.; Chen, B.; Duan, C. Appl. Environ. Microbiol. 2015, 81, 2506.
doi: 10.1128/AEM.04023-14 |
| [33] |
Li, Q.; Sun, B.; Chen, J. Acta Biochim. Biophys. Sin. 2021, 53, 620.
doi: 10.1093/abbs/gmab036 |
| [34] |
Song, C.; Luan, J.; Cui, Q. ACS Synth. Biol. 2019, 8, 137.
doi: 10.1021/acssynbio.8b00402 |
| [35] |
Alberti, F.; Corre, C. Nat. Prod. Rep. 2019, 36, 1237.
doi: 10.1039/C8NP00081F |
| [36] |
Cobb, R. E.; Wang, Y.; Zhao, H. ACS Synth. Biol. 2015, 4, 723.
doi: 10.1021/sb500351f |
| [37] |
Zhang, M. M.; Wong, F. T.; Wang, Y. Nat. Chem. Biol. 2017, 13, 607.
doi: 10.1038/nchembio.2341 |
| [38] |
Huang, H.; Zheng, G.; Jiang, W. Acta Biochim. Biophys. Sin. 2015, 47, 231.
doi: 10.1093/abbs/gmv007 |
| [39] |
Tong, Y.; Charusanti, P.; Zhang, L. ACS Synth. Biol. 2015, 4, 1020.
doi: 10.1021/acssynbio.5b00038 |
| [40] |
Zeng, H.; Wen, S.; Xu, W. Appl. Microbiol. Biotechnol. 2015, 99, 10575.
doi: 10.1007/s00253-015-6931-4 pmid: 26318449 |
| [41] |
Mo, J.; Wang, S.; Zhang, W. Synth. Syst. Biotechnol. 2019, 4, 86.
|
| [42] |
Liu, G.; Chater, K. F.; Chandra, G. Microbiol. Mol. Biol. Rev. 2013, 77, 112.
doi: 10.1128/MMBR.00054-12 |
| [43] |
Bikard, D.; Jiang, W.; Samai, P. Nucleic Acids Res. 2013, 41, 7429.
doi: 10.1093/nar/gkt520 pmid: 23761437 |
| [44] |
Qi, L. S.; Larson, M. H.; Gilbert, L. A. Cell 2013, 152, 1173.
doi: 10.1016/j.cell.2013.02.022 |
| [45] |
Gilbert, L. A.; Larson, M. H.; Morsut, L. Cell 2013, 154, 442.
doi: 10.1016/j.cell.2013.06.044 pmid: 23849981 |
| [46] |
Mali, P.; Aach, J.; Stranges, P. B. Nat. Biotechnol. 2013, 31, 833.
doi: 10.1038/nbt.2675 |
| [47] |
Dong, C.; Fontana, J. Nat. Commun. 2018, 9, 2489.
doi: 10.1038/s41467-018-04901-6 |
| [48] |
Zhang, X.; Tee, L.; Wang, X. Mol. Ther.-Nucleic Acids 2015, 4, e264.
doi: 10.1038/mtna.2015.37 |
| [49] |
Su, Q.; Cheng, C.; Niu, J. Curr. Opin. Chem. Biol. 2021, 64, 10.
doi: 10.1016/j.cbpa.2021.02.004 |
| [1] | 丁卫忠, 张炳文, 薛彦青, 林雨琦, 汤志军, 王婧, 杨文超, 王晓峰, 刘文. 禾谷镰刀菌中一个新的聚酮类化合物[J]. 有机化学, 2023, 43(9): 3319-3322. |
| [2] | 吴秀蓉, 肖朝江, 沈怡, 汤红霞, 朱俊逸, 姜北. 植物来源抗疟倍半萜类天然产物研究(1972~2022)[J]. 有机化学, 2023, 43(8): 2764-2789. |
| [3] | 张怀远, 许诺, 唐蓉萍, 石星丽. 手性高价碘试剂诱导的不对称去芳构化反应研究进展[J]. 有机化学, 2023, 43(11): 3784-3805. |
| [4] | 姚良才, 桂敬汉. 自由基多烯环化反应在天然产物全合成中的应用[J]. 有机化学, 2022, 42(9): 2703-2714. |
| [5] | 孔祥凯, 张逸鹏, 党菱婧, 陈文, 张洪彬. 吲哚生物碱Vindoline与Vindorosine的合成研究进展[J]. 有机化学, 2022, 42(9): 2728-2744. |
| [6] | 高冉, 田伟生. 苦楝甾醇及2α,3α,20R-三羟基孕甾-16β-甲基丙烯酸酯的合成[J]. 有机化学, 2022, 42(8): 2521-2526. |
| [7] | 石发胜, 王圣文, 徐欢, 路星星, 杨新玲, 孙腾达, 王长凯, 张晓鸣, 杨青, 凌云. 新型缩氨基硫脲类化合物的设计、合成及杀菌活性研究[J]. 有机化学, 2022, 42(7): 2106-2116. |
| [8] | 颜雅倩, 王浩鑫, 李瑶瑶. 新多环特特拉姆酸大环内酰胺3-Hydroxycombamide I的发现[J]. 有机化学, 2022, 42(5): 1557-1561. |
| [9] | 罗洁, 颜雅倩, 王浩鑫, 李瑶瑶. 新多环大环内酰胺Clifednamide K的发现[J]. 有机化学, 2022, 42(4): 1224-1228. |
| [10] | 徐萌萌, 蔡泉. 2-吡喃酮的催化不对称Diels-Alder反应研究进展[J]. 有机化学, 2022, 42(3): 698-713. |
| [11] | 马文静, 朱礼志, 章梦珣, 李志成. ent-Kaurene全碳骨架中AB环系的不对称合成[J]. 有机化学, 2022, 42(2): 580-589. |
| [12] | 李泉城, 姜岚, 白瑞, 韩永康, 李争宁. 3-取代苯酞的合成研究进展[J]. 有机化学, 2021, 41(9): 3390-3399. |
| [13] | 陈伟, 刘强. 烯醇类化合物氧化偶联反应研究进展[J]. 有机化学, 2021, 41(9): 3414-3430. |
| [14] | 赵军, 肖检, 王雅雯, 彭羽. 基于氧化[3+2]环加成反应合成二氢苯并呋喃类天然产物[J]. 有机化学, 2021, 41(8): 2933-2945. |
| [15] | 肖检, 彭羽, 李卫东. 倍半萜生物碱Dendrobine的全合成研究进展[J]. 有机化学, 2021, 41(7): 2636-2649. |
| 阅读次数 | ||||||
|
全文 |
|
|||||
|
摘要 |
|
|||||